- Biblioteca de Soluções da AWS›
- Orientações para a integração e análise de dados multiômicos e multimodais na AWS
Orientações para a integração e análise de dados multiômicos e multimodais na AWS
Visão geral
Como funciona
Arquitetura
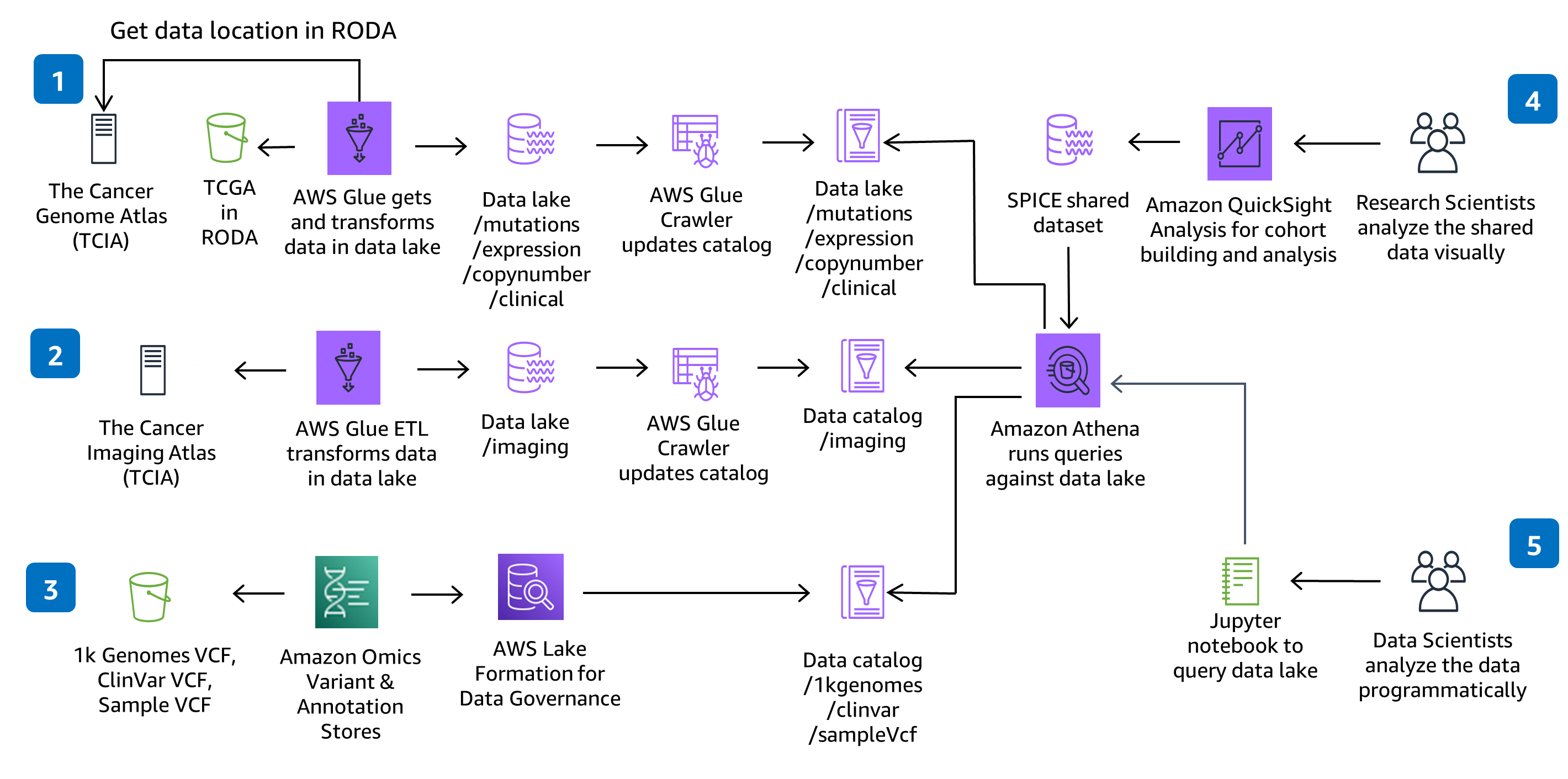
Prepare dados genômicos, clínicos, de mutação, expressão e imagem para análise em grande escala e consulta em um data lake.
CI/CD
Prepare dados genômicos, clínicos, de mutação, expressão e imagem para análise em grande escala e consulta em um data lake.
Pilares do Well-Architected
O diagrama de arquitetura acima exemplifica a criação de uma solução pautada nas melhores práticas do Well-Architected. Para ser totalmente Well-Architected, é preciso respeitar a maior quantidade possível das melhores práticas desse framework.
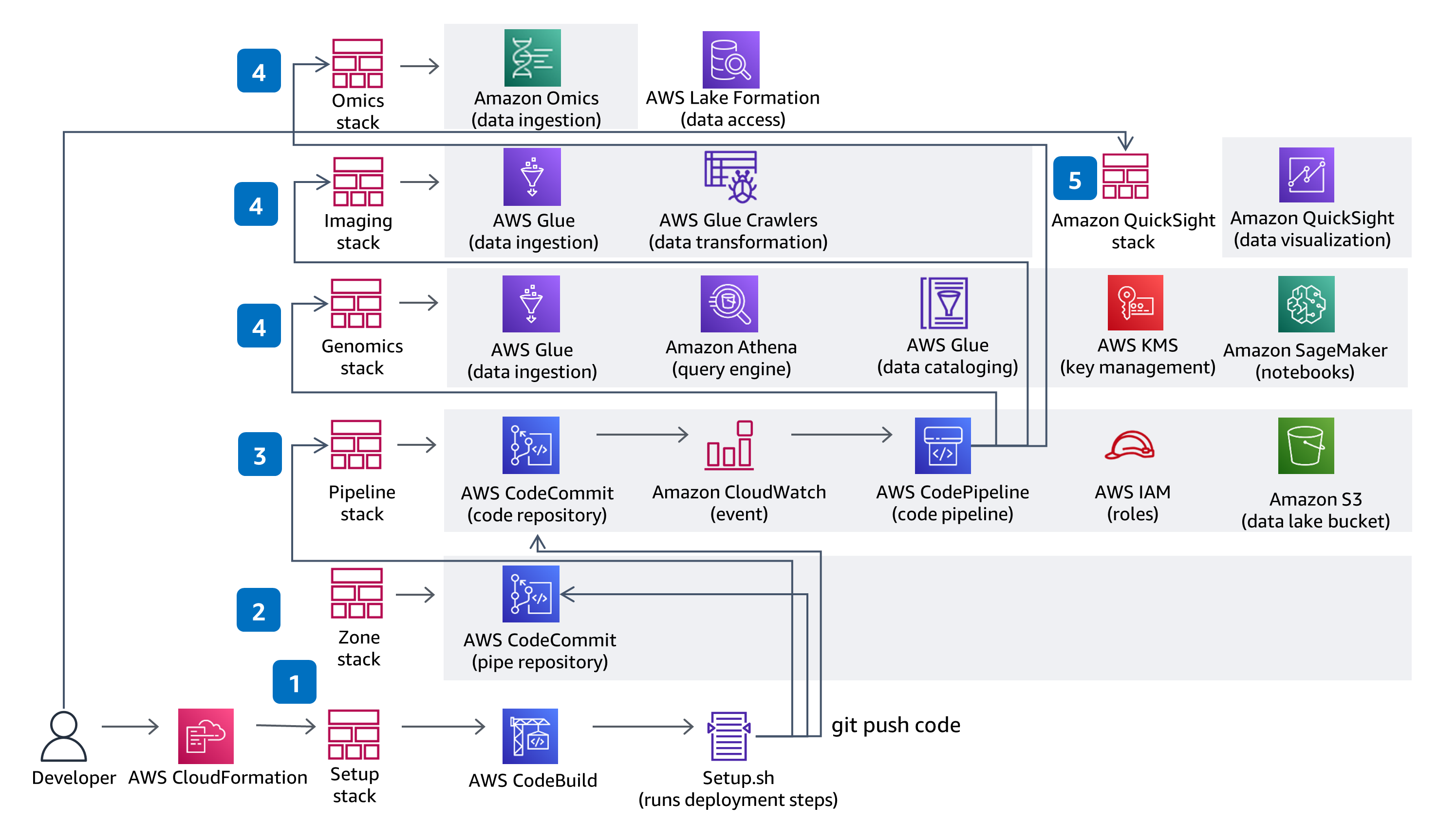
Esta orientação usa o CodeBuild e o CodePipeline para criar, empacotar e implantar tudo o que é necessário na solução para ingerir e armazenar arquivos de chamadas variantes (VCFs) e trabalhar com dados multimodais e multiômicos dos conjuntos de dados do The Cancer Genome Atlas (TCGA) e do The Cancer Imaging Atlas (TCIA). A ingestão e a análise de dados genômicos sem servidor são demonstradas usando um serviço totalmente gerenciado - o Amazon Omics. As alterações de código feitas no repositório da solução CodeCommit devem ser implantadas por meio do pipeline de implantação do CodePipeline fornecido.
Esta orientação usa o acesso baseado em funções com o IAM e todos os buckets têm criptografia habilitada, são privados e bloqueiam o acesso público. O catálogo de dados no AWS Glue tem criptografia habilitada e todos os metadados gravados pelo AWS Glue no Amazon S3 são criptografados. Todas as funções são definidas com privilégio mínimo e todas as comunicações entre os serviços permanecem na conta do cliente. Os administradores podem controlar o notebook Jupyter, os dados do Amazon Omics Variant Store e o acesso aos dados do AWS Glue Catalog é totalmente gerenciado usando o Lake Formation, e o acesso aos dados do Athena, do SageMaker Notebook e do QuickSight é gerenciado por meio das funções do IAM fornecidas.
O AWS Glue, o Amazon S3, o Amazon Omics e o Athena não têm servidor e escalarão o desempenho do acesso aos dados à medida que seu volume de dados aumenta. O AWS Glue provisiona, configura e escala os recursos necessários para executar seus trabalhos de integração de dados. O Athena não tem servidor, então você pode consultar seus dados rapidamente sem precisar configurar e gerenciar servidores ou data warehouses. O armazenamento em memória QuickSight SPICE escalará sua exploração de dados para milhares de usuários.
Ao usar tecnologias sem servidor, você provisiona apenas os recursos exatos que usa. Cada trabalho do AWS Glue provisionará um cluster Spark sob demanda para transformar dados e desprovisionar os recursos quando concluído. Se você optar por adicionar novos conjuntos de dados do TCGA, poderá adicionar novos trabalhos do AWS Glue e crawlers do AWS Glue que também preverão recursos sob demanda. O Athena executa automaticamente as consultas em paralelo, então a maioria dos resultados retorna em segundos. O Amazon Omics otimiza o desempenho de consultas variantes em grande escala ao transformar arquivos no Apache Parquet.
Ao usar tecnologias sem servidor que podem ser escaladas sob demanda, você paga apenas pelos recursos que usa. Para otimizar ainda mais os custos, você pode interromper os ambientes de notebook no SageMaker quando eles não estiverem em uso. O painel do QuickSight também é implantado por meio de um modelo separado do CloudFormation, portanto, se você não pretende usar o painel de visualização, pode optar por não implantá-lo para economizar custos. O Amazon Omics otimiza o custo de armazenamento de dados variantes em grande escala. Os custos de consulta são determinados pela quantidade de dados digitalizados pelo Athena e podem ser otimizados ao escrever consultas adequadamente.
Ao usar amplamente os serviços gerenciados e a escalabilidade dinâmica, você minimiza o impacto ambiental dos serviços de back-end. Um componente essencial para a sustentabilidade é maximizar o uso de instâncias de servidor de blocos de anotações. Você deve interromper os ambientes de notebook quando não estiverem em uso.
Considerações adicionais
Transformação de dados
Essa arquitetura escolheu o AWS Glue para o ETL (Extract, Transform and Load) necessário para ingerir, preparar e catalogar os conjuntos de dados na solução para consulta e desempenho. Você pode adicionar novos AWS Glue Jobs e AWS Glue Crawlers para ingerir novos conjuntos de dados The Cancer Genome Atlas (TCGA) e The Cancer Image Atlas (TCIA), conforme necessário. Você também pode adicionar novos trabalhos e crawlers para ingerir, preparar e catalogar seus próprios conjuntos de dados proprietários.
Análise de dados
Essa arquitetura escolheu os notebooks SageMaker para fornecer um ambiente de notebook Jupyter para análise. Você pode adicionar novos blocos de anotações ao ambiente existente ou criar novos ambientes. Se você preferir o RStudio aos notebooks Jupyter, você pode usar o RStudio no Amazon SageMaker.
Visualização de dados
Essa arquitetura escolheu o QuickSight para fornecer painéis interativos para visualização e exploração de dados. A configuração do painel do QuickSight é feita por meio de um modelo separado do CloudFormation, portanto, se você não pretende usar o painel, não precisa provisioná-lo. No QuickSight, você pode criar sua própria análise, explorar filtros ou visualizações adicionais e compartilhar conjuntos de dados e análises com colegas.
Implemente com confiança
Esse repositório cria um ambiente escalável na AWS para preparar dados genômicos, clínicos, de mutação, de expressão e de imagem para realizar análises em grande escala e consultas interativas em um data lake. A solução demonstra como 1) usar o HealthOmics Variant Store e o Annotation Store para armazenar dados de variantes genômicas e dados de anotação, 2) provisionar canais de ingestão de dados sem servidor para preparação e catalogação de dados multimodais, 3) visualizar e explorar dados clínicos por meio de uma interface interativa e 4) executar consultas analíticas interativas em um data lake multimodal usando o Amazon Athena e o Amazon SageMaker.
Com sua conta da AWS, um guia detalhado é fornecido para experimentação e uso. Cada etapa da criação das orientações, incluindo implantação, uso e limpeza, é examinada para prepará-las para a implantação.
O código de exemplo é um ponto de partida. Ele é validado para o setor, é prescritivo, mas não definitivo, e mostra o que há por trás de tudo para ajudar você a começar.
Conteúdo relacionado
Orientação
Guidance for Multi-Modal Data Analysis with Health AI and ML Services on AWS
Esta orientação demonstra como configurar um framework completo para analisar dados multimodais de saúde e ciências biológicas (HCLS).
Colaboradores

Aviso de isenção de responsabilidade
Você encontrou o que estava procurando hoje?
Informe-nos para que possamos melhorar a qualidade do conteúdo em nossas páginas