AWS 기술 블로그
Strands Agents와 MCP를 사용한 신약 개발 연구 어시스턴트 개발
신약 개발은 복잡하고 시간이 많이 소요되는 과정으로, 연구자들이 방대한 양의 과학 문헌, 임상시험 데이터, 분자 데이터베이스를 탐색해야 합니다. Genentech과 AstraZeneca 같은 생명과학 고객들은 AI 에이전트와 기타 생성형 AI 도구를 사용하여 과학적 발견의 속도를 높이고 있습니다. 이러한 조직의 개발자들은 이미 Amazon Bedrock Agents의 완전 관리형 기능을 사용하여 초기 약물 타겟 식별부터 의료진 참여에 이르기까지 모든 영역에서 도메인별 워크플로우를 신속하게 배포하고 있습니다.
하지만 더 복잡한 사용 사례에서는 오픈소스 Strands Agents SDK를 사용하는 것이 더 유리할 수 있습니다. Strands Agents는 AI 에이전트를 개발하고 실행하기 위한 모델 기반 접근 방식을 취합니다. 커스텀/내부 대규모 언어 모델(LLM) 게이트웨이를 포함한 대부분의 모델 제공업체와 호환되며, Python 애플리케이션을 호스팅할 수 있는 곳 어디서든 에이전트를 배포할 수 있습니다.
이 포스트에서는 Strands Agents와 Amazon Bedrock을 사용하여 신약 개발을 위한 강력한 연구 어시스턴트를 만드는 방법을 보여드립니다. 이 AI 어시스턴트는 모델 컨텍스트 프로토콜(MCP)을 사용하여 여러 과학 데이터베이스를 동시에 검색하고, 발견한 내용을 종합하여 약물 타겟, 질병 메커니즘, 치료법에 대한 포괄적인 보고서를 생성할 수 있습니다.
솔루션 개요
이 솔루션은 Strands Agents를 사용하여 고성능 파운데이션 모델(FM)을 arXiv, PubMed, ChEMBL과 같은 일반적인 생명과학 데이터 소스와 연결합니다. 모델 컨텍스트 프로토콜(MCP) 서버를 신속하게 생성하여 데이터를 쿼리하고 대화형 인터페이스에서 결과를 확인하는 방법을 보여줍니다.
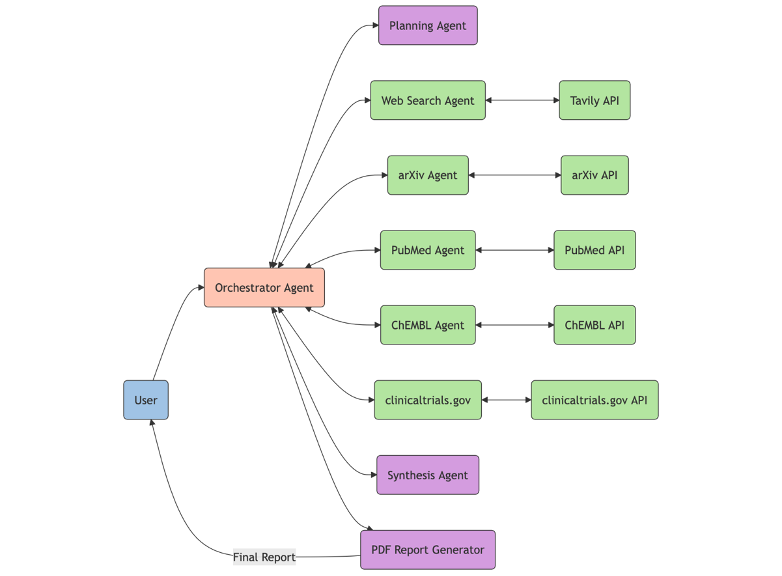
함께 협업하는 전문 AI 에이전트들은 단일한 거대 에이전트보다 종종 더 나은 결과를 만들어낼 수 있습니다. 이 솔루션은 각각 고유한 FM, 명령어, 도구를 가진 하위 에이전트들의 “팀”을 사용합니다. 다음 플로우차트는 오케스트레이터 에이전트(주황색으로 표시)가 사용자 쿼리를 처리하고 정보 검색(녹색) 또는 계획, 종합 및 보고서 생성(보라색)을 위해 하위 에이전트들로 라우팅하는 방법을 보여줍니다.
이 포스트는 로컬 개발 환경에서 Strands Agents로 구축하는 것에 중점을 둡니다. AWS Lambda, AWS Fargate, Amazon EKS 또는 Amazon EC2에서 프로덕션 에이전트를 배포하려면 Strands Agents 문서를 참조하시기 바랍니다.
전제조건
이 솔루션은 Python 3.10+, strands-agents 및 여러 추가 Python 패키지가 필요합니다. 이러한 종속성을 관리하기 위해 venv나 uv와 같은 가상 환경을 사용하는 것을 강력히 권장합니다.
다음 단계에 따라 로컬 환경에 솔루션을 배포하세요.
1. GitHub에서 코드 리포지토리를 클론합니다. 해당 리포지토리에는 다양한 주제의 코드들이 존재하므로 필요한 특정 폴더만 현재 작업 경로에 복사하고 나머지는 삭제하는 방법을 사용했습니다.
git clone https://github.com/aws-samples/aws-ai-ml-workshop-kr.git
cp -r aws-ai-ml-workshop-kr/genai/aws-gen-ai-kr/20_applications/25_drug_discovery_agent ./
rm -rf aws-ai-ml-workshop-kr
cd 25_drug_discovery_agent2. uv가 없는 환경이라면 아래 과정을 통해 설치를 진행합니다.
# 1. uv 설치
curl -LsSf https://astral.sh/uv/install.sh | sh
# 2. 쉘 자동완성 설정 (bash 사용자의 경우)
if [ "$SHELL" = "/bin/bash" ]; then
echo 'eval "$(uv generate-shell-completion bash)"' >> ~/.bashrc
echo 'eval "$(uvx --generate-shell-completion bash)"' >> ~/.bashrc
fi
# 3. 설정 적용
source ~/.bashrc
# 4. uv 최신 버전으로 업데이트
uv self update3. 가상 환경을 구성 후 필요한 Python 종속성을 설치합니다.
uv venv
source .venv/bin/activate
uv pip install -r requirements.txt4. AWS 자격 증명을 환경 변수로 설정하거나, 자격 증명 파일에 추가하거나, 기타 지원되는 프로세스를 따라 구성합니다. 아래 코드는 자격 증명 파일에 프로파일을 추가하는 방법입니다. my-project 대신 원하는 프로파일명을 사용하고, 충분한 권한이 있는 IAM사용자의 AWS Access Key ID와 AWS Secret Access Key를 발급 받아 입력합니다. 이 블로그에서는 Default region name을 us-west-2로 진행합니다.
aws configure --profile my-project다음 명령어를 통해 프로파일을 전환할 수 있습니다.
export AWS_PROFILE=my-project5. 아래 코드를 활용하여 Tavily API 키를 .env 파일에 저장합니다.
- Tavily 홈페이지를 접속하여 가입 후 API Key를 발급할 수 있으며, 무료 버전의 경우 1,000 API Credits/월 이므로 충분히 테스트를 진행할 수 있습니다.
- 아래 코드에서 발급 받은 API Key로 수정한 뒤 코드를 실행합니다.
cat > .env << EOF
TAVILY_API_KEY="YOUR_API_KEY"
EOF6. AWS 계정에서 다음 Amazon Bedrock Foundation Models에 대한 액세스 권한이 필요합니다:
AWS 관리 콘솔 로그인 후 Amazon Bedrock console에 진입 후 좌측 메뉴 하단에 모델 액세스(Model access)를 클릭한 뒤, 위 네가지 모델 접근 권한을 활성화 합니다.
• Anthropic Claude 4 Sonnet
• Anthropic Claude 3.7 Sonnet
• Anthropic Claude 3.5 Sonnet
• Anthropic Claude 3.5 Haiku
7. 한국어로 원활하게 PDF를 생성하기 위해 한국어를 지원하는 폰트(예. 나눔고딕 폰트)의 ttf 파일을 다운로드하여 assets/로 이동하고 chat.py의 font_path를 변경해야 합니다.
절차
파운데이션 모델, MCP 도구, 하위 에이전트를 정의하여 Strands에서 연구 어시스턴트를 생성합니다. 이미 코드는 완성 되어 있으므로 작성된 코드의 핵심 부분만 살펴보겠습니다.
파운데이션 모델 정의
Strands Agents BedrockModel 클래스를 사용하여 Amazon Bedrock의 파운데이션 모델에 대한 연결을 정의하는 것부터 시작합니다. 기본 모델로 Anthropic Claude 3.7 Sonnet을 사용합니다.
[application/chat.py 코드 일부]
from strands import Agent, tool
from strands.models import BedrockModel
from strands.agent.conversation_manager import SlidingWindowConversationManager
from strands.tools.mcp import MCPClient
# Model configuration with Strands using Amazon Bedrock's foundation models
def get_model():
model = BedrockModel(
boto_client_config=Config(
read_timeout=900,
connect_timeout=900,
retries=dict(max_attempts=3, mode="adaptive"),
),
model_id="us.anthropic.claude-3-7-sonnet-20250219-v1:0",
max_tokens=64000,
temperature=0.1,
top_p=0.9,
additional_request_fields={
"thinking": {
"type": "disabled" # Can be enabled for reasoning mode
}
}
)
return modelMCP 도구 정의
MCP는 AI 애플리케이션이 외부 환경과 상호작용하는 방법에 대한 표준을 제공합니다. 생명과학 도구와 데이터셋을 위한 것들을 포함하여 이미 수천 개의 MCP 서버가 존재합니다. 이 솔루션은 다음을 위한 예시 MCP 서버를 제공합니다:
• arXiv: 학술 논문의 오픈 액세스 리포지토리
• PubMed: 생의학 문헌에 대한 동료 심사 인용문헌
• ChEMBL: 약물 유사 특성을 가진 생리활성 분자의 큐레이션된 데이터베이스
• ClinicalTrials.gov: 임상 연구에 대한 미국 정부 데이터베이스
• Tavily Web Search: 공개 인터넷에서 최신 뉴스 및 기타 콘텐츠를 찾기 위한 API
Strands Agents는 에이전트를 위한 MCP 클라이언트 정의를 간소화합니다. 이 예시에서는 표준 입출력을 사용하여 각 도구에 연결합니다. 참고로, Strands Agents는 스트리밍 가능한 HTTP 이벤트 전송을 통한 원격 MCP 서버도 지원합니다.
[application/chat.py 코드 일부]
# MCP Clients for various scientific databases
tavily_mcp_client = MCPClient(lambda: stdio_client(
StdioServerParameters(command="python", args=["application/mcp_server_tavily.py"])
))
arxiv_mcp_client = MCPClient(lambda: stdio_client(
StdioServerParameters(command="python", args=["application/mcp_server_arxiv.py"])
))
pubmed_mcp_client = MCPClient(lambda: stdio_client(
StdioServerParameters(command="python", args=["application/mcp_server_pubmed.py"])
))
chembl_mcp_client = MCPClient(lambda: stdio_client(
StdioServerParameters(command="python", args=["application/mcp_server_chembl.py"])
))
clinicaltrials_mcp_client = MCPClient(lambda: stdio_client(
StdioServerParameters(command="python", args=["application/mcp_server_clinicaltrial.py"])
))전문화된 하위 에이전트 정의
계획 에이전트는 사용자 질문을 살펴보고 어떤 하위 에이전트와 도구를 사용할지에 대한 계획을 수립합니다.
[application/chat.py 코드 일부]
@tool
def planning_agent(query: str) -> str:
"""
A specialized planning agent that analyzes the research query and determines
which tools and databases should be used for the investigation.
"""
planning_system = """
You are a specialized planning agent for drug discovery research. Your role is to:
1. Analyze research questions to identify target proteins, compounds, or biological mechanisms
2. Determine which databases would be most relevant (Arxiv, PubMed, ChEMBL, ClinicalTrials.gov)
3. Generate specific search queries for each relevant database
4. Create a structured research plan
"""
model = get_model()
planner = Agent(
model=model,
system_prompt=planning_system,
)
response = planner(planning_prompt)
return str(response)마찬가지로, 통합 에이전트는 여러 출처의 발견 사항을 하나의 포괄적인 보고서로 통합합니다.
[application/chat.py 코드 일부]
@tool
def synthesis_agent(research_results: str) -> str:
"""
Specialized agent for synthesizing research findings into a comprehensive report.
"""
system_prompt = """
You are a specialized synthesis agent for drug discovery research. Your role is to:
1. Integrate findings from multiple research databases
2. Create a comprehensive, coherent scientific report
3. Highlight key insights, connections, and opportunities
4. Organize information in a structured format:
- Executive Summary (300 words)
- Target Overview
- Research Landscape
- Drug Development Status
- References
"""
model = get_model()
synthesis = Agent(
model=model,
system_prompt=system_prompt,
)
response = synthesis(synthesis_prompt)
return str(response)오케스트레이션 에이전트 정의
또한 전체 연구 워크플로우를 조정하기 위한 오케스트레이션 에이전트를 정의합니다. 이 에이전트는 Strands Agents의 SlidingWindowConversationManager 클래스를 사용하여 대화에서 마지막 10개 메시지를 저장합니다.
[application/chat.py 코드 일부]
def create_orchestrator_agent(
history_mode,
tavily_client=None,
arxiv_client=None,
pubmed_client=None,
chembl_client=None,
clinicaltrials_client=None,
):
system = """
You are an orchestrator agent for drug discovery research. Your role is to coordinate a multi-agent workflow:
1. COORDINATION PHASE:
- For simple queries: Answer directly WITHOUT using specialized tools
- For complex research requests: Initiate the multi-agent research workflow
2. PLANNING PHASE:
- Use the planning_agent to determine which databases to search and with what queries
3. EXECUTION PHASE:
- Route specialized search tasks to the appropriate research agents
4. SYNTHESIS PHASE:
- Use the synthesis_agent to integrate findings into a comprehensive report
- Generate a PDF report when appropriate
"""
# Aggregate all tools from specialized agents and MCP clients
tools = [planning_agent, synthesis_agent, generate_pdf_report, file_write]
# Dynamically load tools from each MCP client
if tavily_client:
tools.extend(tavily_client.list_tools_sync())
# ... (similar for other clients)
conversation_manager = SlidingWindowConversationManager(
window_size=10, # Maintains context for the last 10 exchanges
)
orchestrator = Agent(
model=model,
system_prompt=system,
tools=tools,
conversation_manager=conversation_manager
)
return orchestrator데모: 최신 유방암 연구 탐색
새로운 어시스턴트를 테스트하려면 먼저 코드 리포지토리를 클론하고 전제조건에서 명시한 패키지들을 설치하세요. 그런 다음 streamlit run application/app.py를 실행하여 채팅 인터페이스를 시작하고 웹 브라우저에서 로컬 URL(일반적으로 http://localhost:8501)을 열 수 있습니다.
다음 이미지는 연구 어시스턴트와의 일반적인 대화를 보여줍니다. 이 예시에서는 어시스턴트에게 “최신 뉴스, 최신 연구, 관련 화합물, 진행 중인 임상시험을 포함하여 HER2에 대한 보고서를 생성해 주세요”라고 요청합니다. 어시스턴트는 먼저 사용 가능한 다양한 도구를 활용하여 포괄적인 연구 계획을 수립합니다. HER2에 대한 최신 뉴스 웹 검색과 PubMed 및 arXiv의 과학 논문부터 시작하기로 결정합니다. 또한 ChEMBL에서 HER2 관련 화합물과 진행 중인 임상시험도 살펴봅니다. 이러한 결과들을 하나의 보고서로 종합하고 인용문헌을 포함한 발견사항의 출력 파일을 생성합니다.
 다음은 생성된 보고서의 일부입니다.
다음은 생성된 보고서의 일부입니다.
“종합 과학 보고서: 유방암 연구 및 치료에서의 HER2
1. 요약
인간 표피성장인자 수용체 2(HER2)는 유방암 연구와 치료 개발에서 계속해서 중요한 표적이 되고 있습니다. 이 보고서는 HER2 생물학과 치료적 접근법에 대한 이해의 중요한 진전을 강조하면서 HER2 분야 전반의 최신 발견사항을 종합합니다. 항체-약물 결합체(ADC)의 등장은 HER2 표적 치료의 패러다임 전환을 나타내며, 트라스투주맙 데룩스테칸(T-DXd, Enhertu)은 초기 및 진행성 질환 환경 모두에서 놀라운 효능을 보여주고 있습니다. DESTINY-Breast11 시험은 고위험 초기 단계 HER2 양성 유방암에서 T-DXd에 이어 표준 치료를 시행했을 때 병리학적 완전 반응률에서 임상적으로 의미있는 개선을 보여주어, 잠재적으로 새로운 치료 패러다임을 확립할 수 있음을 입증했습니다.”
중요한 점은 이 작업을 위해 단계별 프로세스를 미리 정의할 필요가 없다는 점입니다. 잘 문서화된 도구 목록만 어시스턴트에게 제공하면, 어시스턴트가 스스로 어떤 도구를 어떤 순서로 사용할지 판단할 수 있습니다.
데모 영상
본 데모에서는 Amazon Bedrock의 Claude Sonnet 4 모델과 MCP(Model Context Protocol)를 활용하여 arXiv, PubMed, ChEMBL, ClinicalTrials.gov 등 핵심 과학 데이터베이스를 실시간으로 통합 분석하는 혁신적인 접근 방식을 확인하실 수 있습니다. 단순한 자연어 질문 입력 만으로, Strands Agent가 복합적 추론을 통해 최적의 데이터 소스를 자동 선택하고, 수집한 정보를 바탕으로 전문가 수준의 분석을 실시간으로 수행합니다. 백엔드 로그를 통해 AI가 어떻게 사고하고 판단하는지 투명하게 확인할 수 있으며, 최종적으로 즉시 활용 가능한 전문가급 PDF 리포트를 자동 생성합니다. 단일 대화형 인터페이스를 통해 표적 단백질의 약물성 평가, 경쟁 파이프라인 분석, 임상 개발 전략 수립 등 실무진이 매일 마주하는 복합적 질의에 대해 기존 수주에서 수개월이 소요되던 연구 작업을 몇 분 내로 완료하는 혁신적 생산성을 직접 확인해 볼 수 있습니다.
정리
로컬 컴퓨터에서 이 예시를 따라하면 정리해야 할 새로운 리소스가 AWS 계정에 생성되지 않습니다. EC2, EKS, Lamdba 등 AWS 컴퓨팅 리소스 중 하나를 사용하여 연구 어시스턴트를 배포하는 경우, 정리 지침은 관련 서비스 문서를 참조하시기 바랍니다.
결론
이 포스트에서는 Strands Agents가 강력하고 도메인별 AI 어시스턴트 생성을 어떻게 간소화하는지 보여드렸습니다. 여러분만의 연구 질문으로 이 솔루션을 시도해보고 새로운 과학 도구로 확장해보시기를 권장합니다.
Strands Agents의 오케스트레이션 기능, 스트리밍 응답, 유연한 구성과 Amazon Bedrock의 강력한 언어 모델의 결합은 AI 지원 연구를 위한 새로운 패러다임을 만들어냅니다. 과학 정보의 양이 계속해서 기하급수적으로 증가함에 따라, Strands Agents와 같은 프레임워크는 신약 개발에 필수적인 도구가 될 것입니다.
Strands Agents로 지능형 에이전트를 구축하는 방법에 대해 자세히 알아보려면 Strands Agents 출시 블로그, 문서, GitHub 리포지토리를 방문하세요. Amazon Bedrock을 기반으로 구축된 헬스케어 및 생명과학용 샘플 에이전트도 찾아보실 수 있습니다.
AWS에서 신약 개발을 위한 AI 기반 솔루션 구현에 대한 자세한 정보는 AWS for Life Sciences에서 확인하세요.