O blog da AWS
Processando 2 petabytes de dados genômicos do TCGA: Como o Hospital Sírio-Libanês acelerou a pesquisa com AWS
Por Gabriela Nova, Arquiteta de Soluções na AWS; Pedro Galante, Pesquisador Sênior no Hospital Sírio Libanês e Thiago Miller, Pesquisador no Hospital Sírio Libanês.
O Hospital Sírio-Libanês (HSL), centro médico acadêmico sem fins lucrativos localizado em São Paulo, é reconhecido internacionalmente por sua excelência em assistência ao paciente, pesquisa translacional e inovação. Com forte atuação em genômica e oncologia, o HSL mantém colaborações com instituições ao redor do mundo para desenvolver soluções que ampliem o entendimento do câncer e impulsionem a medicina de precisão.
Em 2025, o HSL concluiu um projeto de caracterização de retrocópias somáticas (retroCNVs) em milhares de genomas tumorais, processando mais de 2 petabytes de dados genômicos provenientes do The Cancer Genome Atlas (TCGA). Utilizando serviços da AWS, a instituição conseguiu analisar 7.317 amostras (3.603 tumorais e 3.714 normais) de mais de 30 tipos tumorais distintos, uma análise que seria impraticável em infraestrutura local.
Contexto Científico: Retrocópias somáticas
O TCGA criado pelo National Cancer Institute e pelo National Human Genome Research Institute, é um projeto que catalogou e padronizou informações moleculares de mais de 11 mil pacientes, distribuídos entre mais de 30 tipos tumorais distintos. Ele reúne dados de sequenciamento em larga escala – incluindo genoma completo, exoma, transcriptoma, metilação e variantes estruturais – gerados segundo protocolos harmonizados, o que o torna um recurso de referência para a comunidade de bioinformática. A consistência e profundidade desses dados tornam o TCGA especialmente valioso para investigações que buscam compreender mecanismos mutacionais pouco explorados, como é o caso das retrocópias somáticas.
Retrocópias, também chamadas de pseudogenes processados, são variantes estruturais produzidas pela retrotransposição mediada pelo elemento LINE-1, o único retrotransposon autonomamente ativo em humanos. Elas surgem quando o maquinário de LINE-1 copia um RNA mensageiro de volta para o DNA, dando origem a duplicatas que preservam exons contínuos e cauda poli-A, mas não contêm introns. Quando inseridas, por meio de um processo denominado retrotransposição somática, em regiões funcionalmente importantes do genoma, tais retrocópias podem afetar genes essenciais e contribuir para a origem do câncer. A literatura já descreve casos de retrotransposição somática associada à instabilidade genômica, à interrupção de genes supressores tumorais e à progressão neoplásica, reforçando a importância de investigar tais eventos de maneira sistemática em genomas de câncer.
Apesar de sua relevância biológica, a ocorrência de retrocópias somáticas (retroCNVs) permanece subexplorada nos estudos de sequenciamento de genoma completo (WGS) de pacientes com câncer. Isso ocorre em grande parte devido às exigências computacionais envolvidas: arquivos de alinhamento no formato BAM do TCGA frequentemente ultrapassam centenas de gigabytes por amostra, e a detecção precisa de assinaturas de retrotransposição requer algoritmos especializados e grande quantidade de memória RAM. Processar milhares de amostras dessa magnitude em infraestrutura local é inviável, tanto pela capacidade de armazenamento limitada quanto pelo tempo de execução acumulado.
Com isso, o HSL propôs analisar inicialmente cerca de sete mil amostras tumorais (e seus respectivos genomas-controle normais) do TCGA utilizando o algoritmo próprio sideRETRO. O uso dos serviços AWS tornou essa iniciativa viável, oferecendo a escalabilidade necessária para lidar com petabytes de dados, além de paralelizar eficientemente as análises e garantir a segurança no acesso aos dados restritos do consórcio.
Desafio
A análise em larga escala dos dados do TCGA revelou um conjunto de desafios naturais para qualquer projeto que lide com WGS em escala populacional. Cada amostra do TCGA pode ultrapassar centenas de gigabytes de dados brutos no formato BAM, contendo milhões de leituras alinhadas ao genoma humano. Quando multiplicado por milhares de amostras, o volume total rapidamente atinge a ordem de petabytes, tornando impraticável qualquer tentativa de processamento local com infraestrutura tradicional. Mesmo centros com clusters próprios enfrentam limitações sérias relacionadas à capacidade de armazenamento, à largura de banda para leitura simultânea, ao gerenciamento de filas de execução e ao tempo total necessário para concluir a análise.
Além da dimensão dos arquivos, o algoritmo sideRETRO exige um fluxo de processamento intenso em CPU e memória, já que a detecção de retrocópias somáticas depende da identificação de padrões sutis de alinhamento, especialmente em regiões repetitivas do genoma. Esses padrões incluem leituras quiméricas, assinaturas de “soft clipping” e pares de leitura discordantes, que precisam ser avaliados com alta precisão para evitar falsos positivos. Executar esse tipo de análise em milhares de genomas demanda paralelização massiva e capacidade de isolar cada execução para garantir reprodutibilidade e controle de versões, o que é algo difícil de escalar em um ambiente local sem comprometer o desempenho.
Outro obstáculo importante foi o tempo. Mesmo com uma infraestrutura robusta, processar sequencialmente milhares de amostras de WGS levaria meses ou anos, o que inviabilizaria o objetivo do projeto de criar rapidamente um catálogo amplo de retroCNVs que pudesse orientar novas hipóteses científicas. A necessidade de baixar dados restritos do TCGA, gerenciar tokens de acesso, armazenar resultados intermediários, recuperar logs e evitar interrupções em análises longas adicionou ainda mais complexidade ao fluxo de trabalho.
Diante desses fatores, a computação em nuvem emergiu como solução essencial devido às suas características de arquitetura elástica, altamente paralelizável e capaz de operar sob demanda, atendendo aos requisitos de escalabilidade, segurança e tempo de processamento. Assim, a nuvem se apresentou não como uma conveniência, mas como uma necessidade para transformar essa análise em um projeto viável dentro de prazos científicos realistas.
Solução
Para viabilizar a análise em larga escala dos dados do TCGA, o HSL construiu uma arquitetura na AWS capaz de oferecer paralelização massiva, segurança no acesso aos dados e reprodutibilidade em cada execução do pipeline. A solução foi baseada nos serviços Amazon EC2, Amazon S3, Amazon ECR, Amazon ECS e AWS Batch, permitindo que cada etapa – do download dos arquivos BAM ao processamento final pelo sideRETRO – fosse totalmente automatizada e escalável sob demanda.
Para atender às exigências computacionais do projeto, as análises foram executadas em instâncias Amazon EC2 r5.2xlarge, escolhidas por oferecerem o melhor custo benefício entre as máquinas otimizadas para memória. Cada instância disponibilizava 8 vCPUs e 64 GB de RAM, suficientes para suportar as etapas mais intensivas do sideRETRO.
O Amazon S3 serviu como eixo central do armazenamento, abrigando todos os dados necessários para a execução e a auditoria do pipeline. A separação lógica da estrutura facilitou o rastreamento, o controle de versão e a recuperação rápida de resultados.
Durante o processamento, cada instância EC2 foi provisionada com 5 TB de armazenamento Amazon EBS gp3, garantindo throughput consistente para leitura e escrita de arquivos BAM, que frequentemente ultrapassam centenas de gigabytes por amostra.
A organização dos dados foi distribuída nas seguintes categorias:
- Dados de Referência: arquivos FASTA, GTF e índices genômicos usados pelo pipeline.
- Arquivos de Controle: manifestos do GDC contendo metadados das amostras e instruções para download dos BAMs.
- Resultados de Análise: arquivos VCF gerados pelo sideRETRO, além de tabelas auxiliares e metadados.
- Logs e Registros: logs detalhados de execução, essenciais para depuração, auditoria e replicação de resultados.
Essa abordagem centralizada permitiu que instâncias EC2 temporárias pudessem ser criadas e destruídas sem perda de dados, mantendo o pipeline resiliente e facilmente escalável.
Segurança e Conteinerização
Como os dados do TCGA possuem acesso controlado, o pipeline precisava garantir compliance e rastreabilidade durante o download e o processamento. O acesso às amostras foi realizado por meio de tokens de autenticação do GDC, renovados conforme a política de uso do consórcio.
A execução das análises foi encapsulada em um ambiente padronizado utilizando Docker, cuja imagem principal foi armazenada no Amazon Elastic Container Registry (ECR). Esse modelo trouxe diversas vantagens técnicas:
- A imagem contém apenas o ambiente base e as dependências essenciais para o sideRETRO.
- Scripts e parâmetros de execução são recuperados dinamicamente do S3, permitindo atualizações contínuas sem necessidade de reconstruir a imagem Docker.
- O isolamento do ambiente garante reprodutibilidade das análises, independentemente do número de instâncias simultâneas.
Esse desenho reduziu custos operacionais e acelerou o processo de manutenção do pipeline.
Orquestração do Pipeline
A execução em larga escala foi coordenada pelo AWS Batch, responsável por criar filas de jobs, atribuir recursos computacionais e gerenciar automaticamente instâncias EC2 e contêineres Docker. Os contêineres foram executados no Amazon Elastic Container Service (ECS), garantindo controle fino sobre o ciclo de vida de cada job.
A orquestração envolveu:
- A leitura dos manifestos no S3,
- Criação de jobs independentes para cada lote,
- Provisionamento automático das instâncias EC2 com a AMI apropriada,
- Execução dos contêineres sideRETRO carregados diretamente do ECR,
- Envio dos arquivos de saída de volta ao S3.
Dessa forma, o pipeline pôde operar de forma totalmente paralela, escalando automaticamente conforme a demanda de processamento.
Fluxo de Processamento
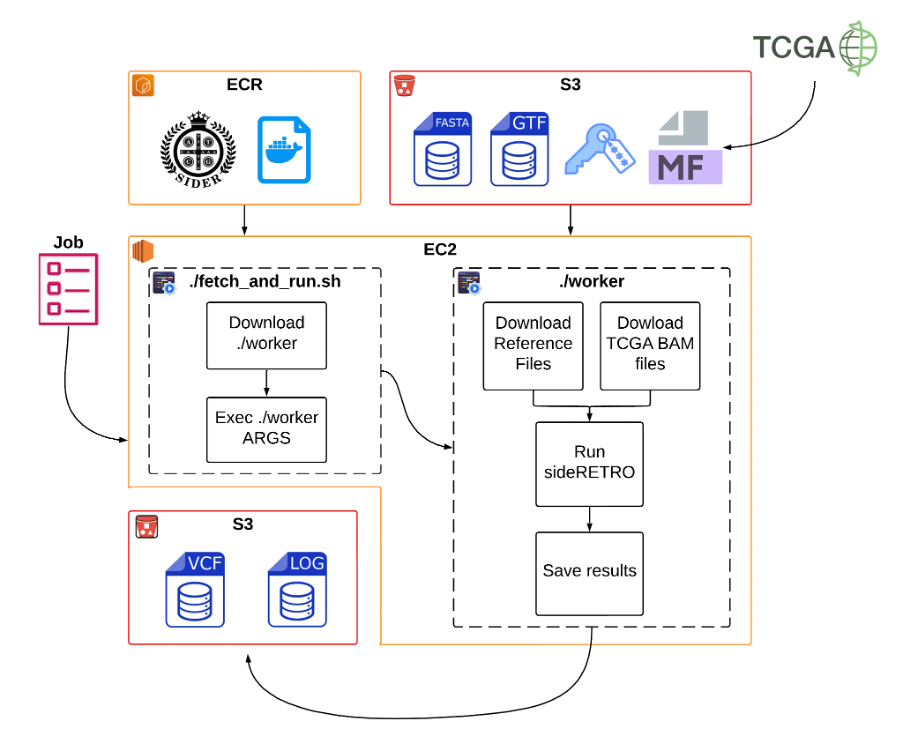
Figura 1. Visão geral da execução do pipeline sideRETRO na AWS.
O pipeline completo (Figura 1) foi estruturado em três grandes etapas, cada uma delas automatizada e integrada aos serviços AWS:
Divisão dos manifestos
O arquivo de manifesto do TCGA foi particionado em lotes menores de dez amostras. Cada lote era empacotado como uma unidade de trabalho independente e gravado no S3.
Processamento por lote
Para cada lote, o AWS Batch iniciava um job, criando dinamicamente uma instância EC2 com os recursos necessários. O contêiner Docker, obtido diretamente do ECR, gerenciava todo o fluxo de execução, recuperando os scripts e os parâmetros armazenados no S3.
Execução e finalização
Cada job realizava o download seguro dos arquivos BAM utilizando o GDC Data Transfer Tool, executava o algoritmo sideRETRO e submetia os arquivos VCF e os logs produzidos de volta ao S3. Ao final da execução, a instância EC2 era encerrada automaticamente, garantindo a redução de custos.
Resultados
A adoção da infraestrutura da AWS viabilizou uma análise de grande escala através de suas capacidades nativas de distribuição e escalabilidade. Ao aproveitar o processamento distribuído entre múltiplas instâncias computacionais e o armazenamento elástico da nuvem, o projeto alcançou um ritmo acelerado de execução, onde os recursos de CPU, memória e espaço em disco se adaptaram dinamicamente às demandas intensivas dos pipelines de bioinformática.
Números do Projeto:
- 2,03 petabytes de dados processados (2.026,76 TB)
- 7.317 amostras analisadas (3.603 tumorais + 3.714 normais)
- 30+ tipos tumorais do TCGA caracterizados
- Modelo de custos: sob demanda, recursos alocados apenas quando necessários
Impacto científico
Do ponto de vista científico, a execução sistemática do sideRETRO sobre os dados de WGS do TCGA permitiu a geração de um catálogo abrangente de retroCNVs em diferentes tipos tumorais. O volume total de dados processados alcançou 2.026,76 TB (~2,03 PB), distribuídos entre 3.603 amostras tumorais e 3.714 amostras normais. Essa escala torna o projeto, até onde sabemos, uma das análises mais amplas já realizadas com foco na identificação de retroCNVs em câncer.
A capacidade de processar em paralelo garantiu que coortes inteiras pudessem ser analisadas de forma consistente e em tempo hábil. Cada execução era realizada em ambientes totalmente reprodutíveis, graças ao uso de contêineres (ECS) e à padronização das dependências. O ciclo operacional também se beneficiou da estrutura de custos sob demanda, com instâncias ativadas apenas durante o processamento. Além disso, todos os artefatos intermediários, logs e resultados finais foram armazenados de maneira segura e versionada, permitindo auditoria, validação independente e reanálises futuras.
“A infraestrutura e o suporte da AWS foram fundamentais para identificarmos esses mecanismos moleculares complexos possivelmente relacionados à origem do câncer, abrindo caminho para futuras estratégias terapêuticas.” Pedro Galante, Pesquisador Sênior no Instituto Sírio-Libanês de Ensino e Pesquisa do Hospital Sírio-Libanês
Conclusão
A implementação deste projeto na AWS demonstrou, de forma clara, como a computação em nuvem pode transformar análises genômicas em larga escala, especialmente quando o objetivo é processar petabytes de dados com reprodutibilidade, segurança e eficiência. Ao combinar serviços como Amazon EC2, Amazon S3, AWS Batch, Amazon ECS e Amazon ECR, o Hospital Sírio-Libanês conseguiu construir uma arquitetura capaz de executar um pipeline complexo e intensivo de bioinformática sem as limitações típicas de ambientes locais.
A possibilidade de escalar horizontalmente o processamento e de orquestrar milhares de tarefas paralelas permitiu que o sideRETRO fosse aplicado de maneira uniforme a todas as amostras do TCGA, resultando na geração de um catálogo abrangente de retrocópias somáticas. A infraestrutura elástica da AWS não apenas acelerou a análise, reduzindo o tempo total de processamento de meses para semanas, como também contribuiu para a otimização de custos, ao garantir que os recursos computacionais fossem utilizados apenas durante as janelas efetivas de execução.
Do ponto de vista científico, o projeto resultou na construção de um recurso valioso para a comunidade de pesquisa em câncer: um catálogo sistemático e de grande escala de retroCNVs, agora pronto para ser disponibilizado publicamente. Esse produto final representa uma ponte entre tecnologia e medicina, ilustrando como abordagens computacionais avançadas podem impulsionar descobertas em oncologia molecular.
AWS Open Data Program
Além da infraestrutura computacional propriamente dita, um componente fundamental do ecossistema da AWS para a pesquisa científica é o programa AWS Open Data, que hospeda conjuntos de dados públicos de grande relevância diretamente em buckets Amazon S3 acessíveis globalmente. Esse modelo reduz substancialmente os custos e a complexidade operacional associados à movimentação de grandes volumes de dados, ao mesmo tempo em que oferece alta largura de banda e integração direta com os serviços de computação em nuvem.
Embora o presente projeto com dados do TCGA não tenha feito uso direto do AWS Open Data, essa infraestrutura já foi essencial em trabalhos anteriores desenvolvidos pelo grupo. Em um estudo voltado à identificação de retroCNVs polimórficas em populações humanas, foram analisados dados da Fase 3 do projeto 1000 Genomes, disponibilizados publicamente pela AWS em um bucket dedicado. Esse conjunto reúne genomas completos de 2.504 indivíduos, sequenciados com cobertura média de 30 vezes e representando 26 populações humanas distintas. O acesso direto aos dados no S3 permitiu que todas as etapas de processamento fossem realizadas inteiramente dentro da infraestrutura da AWS, com altas taxas de transferência e sem os custos associados à movimentação de dados.
A colaboração entre o Hospital Sírio-Libanês e a AWS reforça a importância de infraestruturas modernas para viabilizar pesquisas de alto impacto. Com essa base estabelecida, novas análises, reanálises e integrações com outros bancos de dados genômicos tornam-se possíveis, abrindo caminho para estudos ainda mais aprofundados sobre a dinâmica da retrotransposição no câncer e suas implicações biológicas.
Saiba mais sobre o AWS Open Data Program para acesso a datasets públicos de genômica
Autores
 |
Gabriela Nova é formada em Análise e Desenvolvimento de Sistemas pelo Instituto Federal de São Paulo (IFSP) e atualmente atua como Arquiteta de Soluções na AWS no setor de saúde. Com cinco anos de experiência em AWS, possui vivência nos setores de saúde e financeiro, e integra a comunidade técnica de Inteligência Artificial da AWS. |
 |
Pedro A. F. Galante é cientista com formação acadêmica iniciada em Ciências Moleculares pela Universidade de São Paulo (USP), seguida de doutorado em Bioquímica e Biologia Molecular pela mesma instituição. Realizou pós-doutorado na Universidade de Harvard, nos Estados Unidos. Atuou como Pesquisador Pleno no Ludwig Institute for Cancer Research e integrou a diretoria da Associação Brasileira de Bioinformática e Biologia Computacional. Atualmente, é Pesquisador Sênior no Instituto Sírio-Libanês de Ensino e Pesquisa e atua como coordenador adjunto do Programa de Pós-graduação em Ciências da Saúde do Hospital Sírio-Libanês. |
 |
Thiago L. A. Miller é formado em Engenharia Biotecnológica pela Universidade Estadual Paulista (UNESP), campus de Assis, e possui doutorado em Ciências pelo Instituto de Química da Universidade de São Paulo (USP). Realizou pós-doutorado no Instituto Sírio-Libanês de Ensino e Pesquisa e, atualmente, é pesquisador de pós-doutorado no Laboratório Nacional de Computação Científica (LNCC).
|