Amazon Web Services ブログ
Amazon Comprehend Medical を使用して、診療記録を OMOP 共通データモデルとヘルスケアオントロジーにマッピングする
看護観察データと共に患者の健康状態を記述できるということは、現代のヘルスケアシステムにおいて重要な側面を持っています。定量化できる個人の健康情報の量は膨大で、新しい健康管理法、基準、デバイスが導入されるに従って、その量は継続的に増えていきます。このデータはいずれも、臨床医や研究員が時間の経過と共に、患者の健康状態がどのように変化するかを把握したり、正確な治療の機会を特定したりするのを可能にします。こうしたデータの集合によって、疫学者は人々の健康状態を知り、因果関係のパターンを特定できるようになるのです。
決まった形のない文字情報、通例、カルテなどの診療記録は、患者の看護観察健康データの豊かな情報源と言えます。形式化された患者の健康状態記録には含まれない重要な情報が、臨床医によって書き込まれることもよくあります。形式化された健康状態記録データの品質評価を支援するために、診療記録が使用されることもあります。これまで診療記録では、そこに含まれる医療的洞察を抽出するために、時間とコストのかかる手作業での検証が必要であることが課題でした。
Amazon Comprehend Medical は健康状態、投薬、服用量、耐久力などの洞察を素早く、正確に抽出するために機械学習を使用する自然言語処理 (NLP) サービスです。お客様は従量課金モデルで Amazon Comprehend Medical を使用し、お客様ご自身で複雑な機械学習モデルを開発したり、トレーニングしたりといった手間をかける必要なく、すぐに医療関連文字情報から洞察を抽出できるようになります。
Observational Health Data Science and Informatics (OHDSI) のコミュニティによって管理されている Observational Medical Outcomes Partnership (OMOP) 共通データモデルは、ヘルスデータに使用される業界標準のオープンソースデータモデルです。OMOP では看護観察健康状態データを格納するために、標準化されたメディカルオントロジー、あるいは、SNOMED などの「単語集」を使用します。出典 OHDSI ウェブサイト:
「OMOP 共通データモデルにより、雑多な看護観察データベースの系統的分析が可能になります。このアプローチの背後にあるコンセプトは、これらのデータベースに含まれるデータを共通のフォーマット (データモデル) と共に、共通の表現 (単語、用語、コーディングスキーマなど)、その後、共通のフォーマットをベースに書かれた標準分析ルーチンのライブラリを使用して体系的な分析することです。」
OMOP の特徴は、雑多なヘルスデータソースから取得された診療記録を格納する機能です。このデータモデルでは、これらの記録は個々の患者と受診を紐付けし、記録をより分かりやすくします。OMOP にはまた、自然言語処理 (NLP) エンジンによって、記録から推測した洞察を格納する機能もあります。本ブログ記事では、OMOP の記録を読み取ったり、医療的洞察を抽出したり、さらには、患者と住民の看護観察健康状態データを強化するため、SNOMED オントロジー用のコードを用いて OMOP に書き込むために、Amazon Comprehend Medical をどのように使用できるかについてご紹介します。
OMOP の記録処理アーキテクチャ
この例では、全 OHDSI アーキテクチャのより大きな規模の診療記録を使って作業します。この GitHub のリポジトリにアクセスすることで、AWS における OHDSI の自動化アーキテクチャについてさらに詳しくご覧いただけます。ここに掲載したコードは特に、OMOP の共有データモデルから記録を読み出し、洞察を同データモデルに書き込みます。しかし、同じ一般的なプロセスは、診療記録に対応する看護観察データ用の他の構造化データモデルと共に、使用することもできます。
OMOP の持つ性能の主な特徴は、多くの地理的な地域から電子カルテ (EHR) システムや管理クレームデータなどの多様な情報源から共通データモデルへデータを集約できることです。OMOP では雑多な情報源から看護観察データを分析できます。それを可能にしているのは、SNOMED、RxNorm、ICD10、Read、LOINC、その他に含まれる多数のソース単語 (辞書またはオントロジーと呼ばれることもあります) を OMOP の標準用語 1 語にマッピングするという手法です。こうして OMOP はある種のロゼッタストーンの役割を果たし、さまざまな情報源と世界の多様な地域からの看護観察データを解釈できるようにします。
ここで実演するアプローチの重要な側面のひとつは、Amazon Comprehend Medical によって検出されたエンティティを、SNOMED オントロジーにマッピングするという手法です。観察結果は OMOP データモデルの標準コードを使用して表現しなければならないため、Amazon Comprehend Medical によって検出された洞察を最初に標準のオントロジーにマッピングしてからでないと、OMOP に書き込むことができません。これはエンティティの文字情報を SNOMED CT Browser サービスへ送信し、各エンティティの SNOMED コードを受け取ることで実現できます。次の図では、このプロセスのアーキテクチャを示しています。
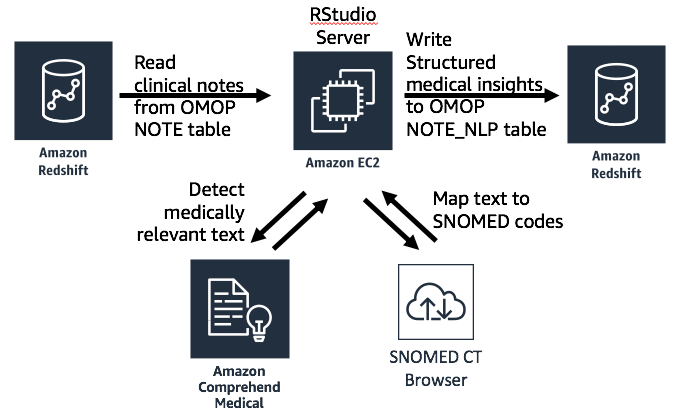
記録の処理に R コードを使用する
GitHub からこのプロセスを実演する R コードをダウンロードできます。
この R コードは RStudio Server Open Source Edition インスタンスを実行する Amazon EC2 インスタンスで稼働中の OHDSI on AWS アーキテクチャの中で実演されています。RStudio は R と連携するウェブベースの統合開発環境 (IDE) です。これは商業的または AGPLv3 の下でラインセンス化できます。
ここで使用されているコードは、Amazon Comprehend Medical をコールするために、AWS 以外の環境からでも使用できます。AWS 外で使用する場合は、このコードが Amazon Comprehend Medical サービスをコールできるようにする認証情報ファイルを構成する必要があります。こちらでその処理に関する説明をご覧いただけます。
このノートの冒頭で、OMOP CDM 接続の詳細とスキーマ名を定義する次のような記述が見られます。OHDSI on AWS アーキテクチャを使用している場合は、これらはホームディレクトリの connectionDetails.R と呼ばれるファイルに含まれています。AWS の外からであれば、ご使用のアーキテクチャに固有の行と共に、これらの行を入力します。
Amazon Comprehend Medical が検出されたすべてのエンティティ、特質、属性についての信頼度を返します。コードの次の処理では、NOTE_NLP テーブルに書き込むために検出機能が満たさねばならない最低限の信頼度スコアを定義する変数が出てきます。この最低限の信頼度スコアはご使用のユースケースに合わせて変更できます。
Amazon Comprehend Medical への実際のコールは、AWS SDK for Python (boto3) を使用して実装されます。R 内から Python コードを実行し、記録の文字情報に渡し、Amazon Comprehend Medical によって検出されたエンティティデータを受け取るために「reticulate」と呼ばれる R ライブラリが使用されます。
OMOP NOTE テーブルで見つかったすべての記録はSQL クエリを使用して読み込まれ、処理されます。Amazon Comprehend Medical によって各記録で検出された各エンティティのために、SNOMED CT Browser を呼び出すことで、適切な SNOMED コードが判別されます。
各記録が処理されるのに従って、R コンソールに情報が出力されます。そこには、NOTE_NLP テーブルに書き込まれた各レコードが表示されます。
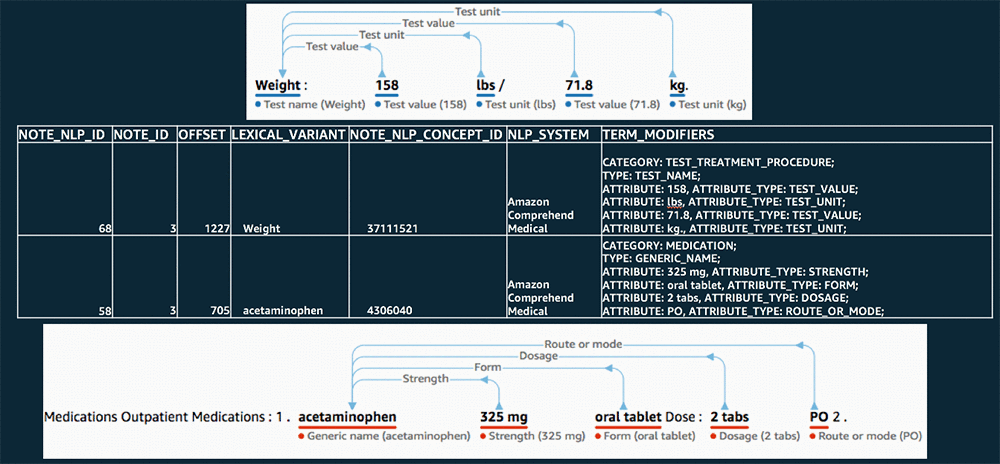
処理が完了すると、NOTE テーブルの記録から抽出された医療的洞察を示すレコードでいっぱいになった NOTE_NLP テーブルが完成します。これらの各レコードには OMOP Standard Concept ID (NOTE_NLP_CONCEPT_ID フィールド) が付与され、検出された主要なエンティティを示す SNOMED コードからのマッピングが作成されています。NOTE_NLP テーブルの TERM_MODIFIERS フィールドは、各エンティティについて Amazon Comprehend Medical が検出したカテゴリ、タイプ、属性データまたは特性データを取得するために使用されます。
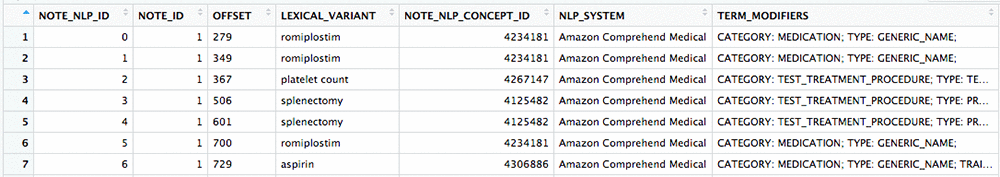
以下の画像はオリジナルの記録に含まれていた文字情報の一部および Amazon Comprehend Medical によって検出されたエンティティと、OMOP に書き込まれた対応する NOTE_NLP レコードとの比較です。
結論
この例では Amazon Comprehend Medical で、医療用の文字情報向け自然言語処理がいかに簡単に利用できるようになるかについてご紹介しています。Amazon Comprehend Medical によって検出された洞察により、データサイエンティスト、疫学者、医療研究者らが、形式化されていない記録に閉じ込められた情報を使って、患者や住民の形式化された看護観察健康レコードを拡大できるようになります。この追加データは正確な投薬、長期的調査、臨床試験の候補者選定、住民の健康に関する研究などに重要な洞察をもたらします。ここで使用されているコードは、OMOP 共通データモデルで医療記録の処理をすぐに開始するのに役立てたり、医療記録を任意の看護観察健康データモデルを処理するためのパターンとして使用したりすることができます。
著者について
 James Wiggins は AWS のシニアヘルスケアソリューションアーキテクトです。 彼は組織が世界の医療にポジティブな影響を与えるようにテクノロジーを使用することに情熱をもっています。彼はまた、妻と3人の子供と余暇を楽しんでいます。
James Wiggins は AWS のシニアヘルスケアソリューションアーキテクトです。 彼は組織が世界の医療にポジティブな影響を与えるようにテクノロジーを使用することに情熱をもっています。彼はまた、妻と3人の子供と余暇を楽しんでいます。