¿Qué le pareció este contenido?
- Aprender
- CelerisTx: descubrimiento de fármacos para enfermedades incurables con ML en AWS
CelerisTx: descubrimiento de fármacos para enfermedades incurables con ML en AWS
Sacar partido del descubrimiento de fármacos mediante computación en compuestos inductores de proximidad (PIC)
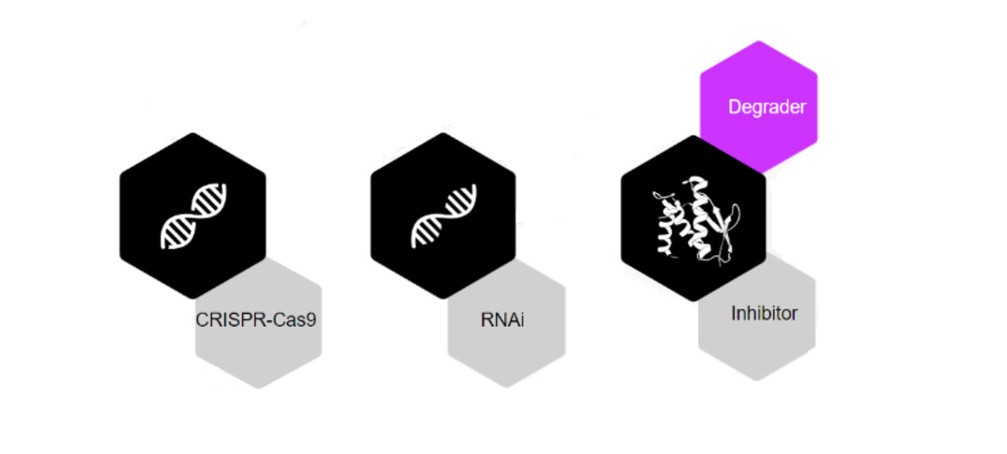
La humanidad ha salido adelante con innumerables soluciones terapéuticas para tratar las enfermedades. Las tecnologías recientes, como el bloqueo (CRISPR, que funciona en el nivel del ADN) y el silenciamiento (RNAi, que funciona en el nivel del ARN) tienen limitaciones importantes. Por el contrario, los compuestos inductores de proximidad (PIC) combinan las ventajas únicas de las moléculas pequeñas. Estas características beneficiosas incluyen la biodisponibilidad oral y la facilidad de fabricación, junto con las de las tecnologías silenciadoras como el CRISPR y el RNAi. A diferencia del RNAi y el CRISPR, los PIC afectan a las proteínas, por lo que constituyen una innovadora modalidad terapéutica para dirigirse a las proteínas asociadas específicamente a las enfermedades. Entre las alternativas que se dirigen a las proteínas se incluyen los compuestos biológicos y los inhibidores y estas alternativas no han ampliado la gama de proteínas patógenas a las que se pueden dirigir. Más del 80 % de las proteínas patógenas están asociadas a una enfermedad que aún no es susceptible de intervención farmacéutica.
La degradación dirigida de proteínas (TPD) es la modalidad de fármacos inductores de proximidad más popular, que se demostró por primera vez a principios de la década de 2000. También recibió el Premio Nobel de Química por descubrir la degradación de proteínas mediada por la ubiquitina en 2004. El concepto de TPD implica la degradación selectiva de proteínas mediante la apropiación de la maquinaria celular interna del cuerpo humano. En comparación con los métodos basados en la ocupación, como los inhibidores, los mecanismos de la TPD tienen la ventaja de eliminar las funciones de andamiaje y, por lo tanto, abordar la raíz del problema, en lugar de limitarse a tratar los síntomas. En general, se necesitan herramientas para eliminar las proteínas mal plegadas y desnaturalizadas de una célula a fin de mantener la homeostasis biológica.
Si bien se pueden utilizar activamente varios mecanismos a través de los PIC, como los lisosomas y la autofagia, Celeris Therapeutics se centra en el sistema ubiquitina-proteasoma. En función de este sistema, las proteínas no deseadas se etiquetan con ubiquitina, una pequeña proteína de señalización. Como resultado, este proceso inicia la degradación de las proteínas patógenas.
Sin embargo, la evaluación de la TPD en un laboratorio experimental es lenta y costosa. Por lo tanto, Celeris Therapeutics implementó un flujo de trabajo de computación para predecir la degradación de las proteínas de manera eficaz y así acortar y agilizar el cronograma de desarrollo de fármacos.
Compuestos inductores de proximidad
Celeris Therapeutics (CelerisTx) lidera la adopción de la IA en los PIC y se centra en la degradación dirigida de proteínas. En términos de datos, en la degradación de proteínas, eso significa identificar múltiples moléculas que interactúan entre sí de una manera particular. Imagine los ladrillos LEGO®. En lugar de unirlos según una sola dimensión, es decir, la forma, estos elementos tienen múltiples dimensiones o requisitos que satisfacer. Algunos son químicos, biológicos, físicos, etc., lo que significa que las interacciones están sujetas a leyes específicas. Estas leyes deben cuantificarse e incorporarse a los datos. Entre los ejemplos concretos de dicha información se incluyen las estructuras tridimensionales de moléculas representadas como grafos, la hidrofobicidad o el potencial electrostático. En total, esto asciende a aproximadamente 20 características diferentes seleccionadas por expertos en TPD. Como resultado, se genera una enorme cantidad de datos que deben analizarse mediante algoritmos de machine learning y, en concreto, canalizaciones de aprendizaje profundo geométrico. Se requiere una aplicación concreta de ML para determinar el grado de interacción entre las moléculas implicadas en el denominado complejo ternario. En otras palabras, si consideramos dos proteínas, esperamos un valor escalar que determine el grado de interacción. Además de ser precisos y generalizables, tenemos que hacer predicciones rápidas en un sistema eficaz. Al simular diferentes interacciones entre proteínas, es necesario descartar rápidamente las predicciones inexactas para analizar el espacio con rapidez. Esto ha demostrado ser un desafío para un conjunto de datos de interacciones con más de 20 000 pares de proteínas.
Cómo se utilizó AWS para varios proyectos de descubrimiento de PIC
Para ahorrar tiempo a los desarrolladores y obtener información más rápida de los problemas específicos relacionados con el desarrollo de machine learning, recurrimos a Amazon SageMaker. Sus capacidades nos permitieron evitar implementar por nuestra cuenta algunas soluciones de infraestructura de machine learning, como la detección de sesgos o el ajuste de hiperparámetros. La detección de sesgos fue importante durante la preparación de los datos; por eso, utilizamos SageMaker Clarify. Esta característica fue fundamental para garantizar la calidad de nuestros datos, incluso antes de empezar a modelar. Posteriormente, una vez que empezamos a modelar, era importante contar con integraciones directas de bibliotecas de aprendizaje profundo geométrico, como la biblioteca Deep Graph Library (DGL) de SageMaker. Dado que DGL es un paquete de Python de código abierto para el aprendizaje profundo en grafos, lo utilizamos para configurar rápidamente la infraestructura necesaria para el aprendizaje profundo geométrico, algo que era esencial.
Una vez configurados los modelos iniciales, para encontrar rápidamente los mejores hiperparámetros, utilizamos el Ajuste automático de modelos de Amazon SageMaker. Estimamos que así evitábamos meses de desarrollo para codificar los marcos de optimización de hiperparámetros. Utilizamos los Experimentos de Amazon SageMaker para hacer un seguimiento de todos los cambios realizados en los experimentos y organizarlos. Esta solución fue especialmente importante. Pensemos en el problema mencionado anteriormente de determinar la intensidad de la interacción entre las proteínas implicadas en el complejo ternario, las más de 15 arquitecturas de aprendizaje profundo geométrico con las que hemos experimentado a partir de capas de atención, los operadores de convolución puros de los grafos, etc. La trazabilidad de los diferentes modelos y sus respectivos parámetros era importante para determinar qué funcionaría en última instancia. Una vez que habíamos creado la versión final del modelo, necesitábamos depurarlo y seguir optimizando la canalización. Para ello, el Depurador de SageMaker resultó útil.
Junto con las soluciones de software de SageMaker, necesitábamos una gran cantidad de potencia de computación para nuestras grandes canalizaciones de datos y aprendizaje profundo. Por ello, creamos una solución de flota de spot optimizada para las GPU utilizadas en el entrenamiento de nuestros flujos de trabajo de machine learning.
Optimizar el costo de nuestra canalización de ML
Las instancias de spot son un tipo especial de instancia de Amazon EC2 que ofrecen un descuento del 90 % en comparación con los precios bajo demanda. A veces, debido a la gran demanda de recursos de computación, estos recursos pueden interrumpirse, lo que hace que el usuario pierda toda una sesión de computación. Es fundamental guardar los resultados intermedios de la computación si se entrena un modelo de aprendizaje profundo durante mucho tiempo o si en la instancia de spot se ejecutó alguna computación necesariamente de larga duración. Además de guardar los resultados intermedios de la computación, el usuario también debería poder continuar automáticamente la computación en otra instancia de spot desde el mismo paso intermedio en el que se produjo la desconexión.
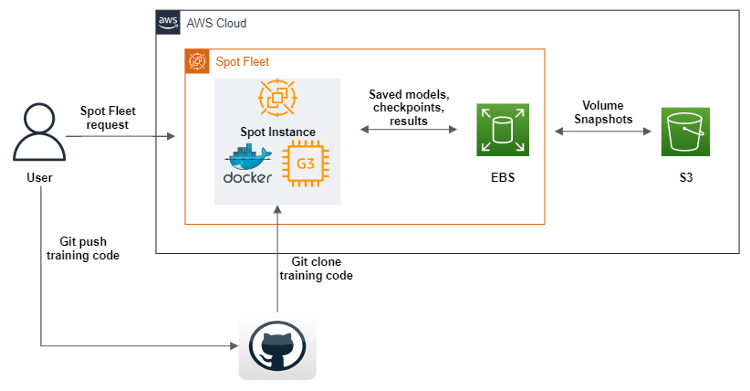
El AWS SDK para Python (Boto3) se usó para crear un script que, tras su ejecución, envía una solicitud de la flota de spot para crear una instancia de spot de GPU con imágenes de máquinas de Amazon (AMI), que contienen todo el software y las dependencias para ejecutar nuestro código de machine learning. Además, definimos scripts de datos de usuario que descargan una imagen de Docker de Amazon Elastic Container Registry (Amazon ECR) y, a continuación, Git clona el código de nuestro repositorio. Por último, los scripts de datos de usuario ejecutan el código de la canalización de aprendizaje profundo geométrico. Este código se ha modificado para que, tras cada etapa de entrenamiento de machine learning, envíe conjuntos de datos, registros, modelos y puntos de control a un volumen de Amazon EBS. Para asegurarnos de que los objetos se guarden, los volcamos todos en Amazon S3. Además de definir una flota de spot, también hemos habilitado la reposición de cualquier instancia de spot que se desconecte durante el proceso, lo que nos permite continuar el entrenamiento automáticamente desde los últimos puntos de control encontrados en los buckets de S3.
SageMaker ofrece algo muy similar, que se conoce como entrenamiento de spot administrado. Sin embargo, no utilizamos la oferta de SageMaker porque, cuando se usan como parte de SageMaker, las instancias de EC2 son más caras que las estándar. Esto tiene sentido, ya que SageMaker ofrece muchas ofertas de ML, es fácil de usar, requiere menos tiempo de ingeniería, etc. Para nosotros, diseñar esta solución fue una inversión estratégica de tiempo, porque sabíamos que íbamos a utilizar las GPU con diferentes capacidades durante varios años.
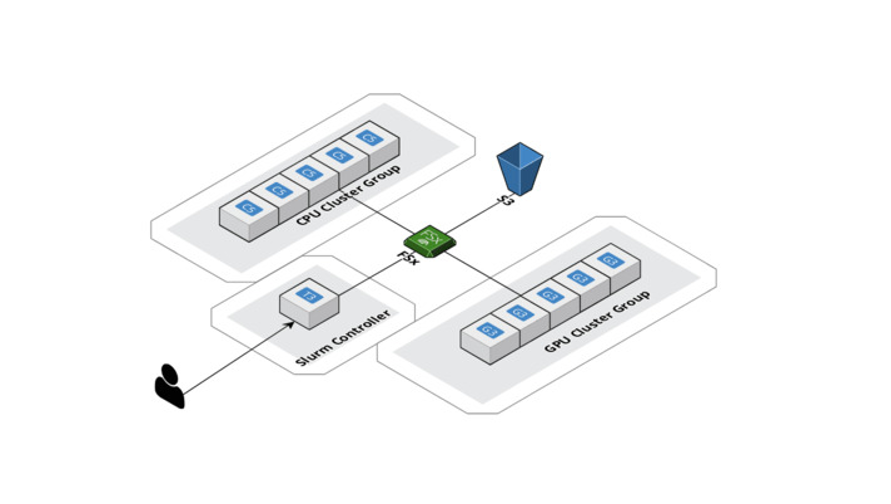
La HPC era otro caso de uso importante que teníamos que atender con AWS. El enorme espacio de datos que debe analizarse mediante el aprendizaje profundo geométrico conlleva una importante carga computacional. Escalamos verticalmente nuestras computaciones con AWS ParallelCluster y creamos un sistema de archivos compartidos, Amazon FSx para Lustre, de modo que todos los nodos de computación en la nube pudieran acceder a los datos y modificarlos desde el mismo lugar. Además, AWS admite Slurm como administrador de cargas de trabajo, lo que nos permite distribuir el trabajo entre las CPU y las GPU al mismo tiempo. A la larga, esto permite no solo una experimentación más rápida, sino también una publicación optimizada de modelos. Una vez más, hay que tener en cuenta que SageMaker ofrece un servicio similar para conjuntos de datos y modelos de gran tamaño, que se conoce como bibliotecas de entrenamiento distribuido.
El futuro de los compuestos inductores de proximidad
Los resultados preliminares de nuestra canalización están disponibles para la progresión de la enfermedad de Parkinson a estudios preclínicos. Experimentar de forma rápida y fiable es fundamental para el descubrimiento de fármacos mediante computación, ya que continuamos mejorando nuestra canalización de descubrimiento de fármacos con nuevas arquitecturas y enfoques. Nuestro enfoque de I+D, en cuya investigación invertimos importantes recursos, implica que nos enfrentamos a muchos riesgos continuos que debemos mitigar. Las ofertas estándar de AWS son una forma de garantizar que somos ágiles y pasamos más rápido de la investigación al laboratorio y, después, al mercado.
Conclusión
Ha llegado el momento de aumentar la productividad en el descubrimiento de fármacos. Lo opuesto a la Ley de Moore en farmacología, que se conoce como “Ley de Eroom”, indica que los costos de investigación y desarrollo necesarios para desarrollar un nuevo fármaco seguirán aumentando. Un enfoque para abordar este problema consiste en utilizar y agilizar el proceso de realización de experimentos de machine learning continuos para el descubrimiento de fármacos.

Christopher Trummer
Christopher Trummer es cofundador de Celeris Therapeutics y se desempeña como director ejecutivo. Ha sido ponente invitado sobre inteligencia artificial en varias ocasiones en conferencias sobre el descubrimiento de fármacos y es coautor de publicaciones revisadas por pares en varias revistas.

Noah Weber
Noah Weber se desempeña como director de tecnología en Celeris Therapeutics. Es Gran Maestro de Kaggle y profesor adjunto en la Universidad de Tecnología de Viena y en la Universidad de Ciencias Aplicadas de Viena.

Olajide Enigbokan
Olajide Enigbokan es Arquitecto de soluciones para startups en Amazon Web Services. Le encanta trabajar con startups (especialmente con desarrolladores) para descubrir el valor de la nube de AWS.

AWS Editorial Team
El equipo de marketing de contenido para startups de AWS colabora con startups de todos los tamaños y sectores para ofrecer contenido excepcional que eduque, entretenga e inspire.
¿Qué le pareció este contenido?