Wie war dieser Inhalt?
- Lernen
- CelerisTx: Wirkstoffforschung für unheilbare Krankheiten mit ML in AWS
CelerisTx: Wirkstoffforschung für unheilbare Krankheiten mit ML in AWS
Computergestützte Arzneimittelforschung für Proximitätsinduzierenden Substanzen (Proximity Inducing Compounds, PICs)
Die Menschheit hat unzählige therapeutische Lösungen zur Behandlung von Krankheiten entwickelt. Neuere Technologien wie Knockout (CRISPR - funktioniert auf DNA-Ebene) und Knockdown (RNAi - funktioniert auf RNA-Ebene) haben erhebliche Beschränkungen. Im Gegensatz dazu vereinen Proximitätsinduzierenden Substanzen (PICs) die einzigartigen Vorteile von kleinen Molekülen. Zu diesen vorteilhaften Eigenschaften gehören die orale Bioverfügbarkeit und eine einfache Herstellung – zusammen mit denen von Silencing-Technologien wie CRISPR und RNAi. Im Gegensatz zu RNAi und CRISPR wirken PICs auf Proteine und stellen damit eine bahnbrechende therapeutische Methode dar, um auf Proteine zu zielen, die speziell mit Krankheiten in Verbindung stehen. Zu den Alternativen, die auf Proteine abzielen, gehören Biologika und Inhibitoren, und diese Alternativen haben das Spektrum der pathogenen Proteine, auf die abgezielt werden kann, nicht erweitert. Mehr als 80 % der pathogenen Proteine werden mit einer Krankheit in Verbindung gebracht, für die es noch keine pharmazeutischen Behandlungsmöglichkeiten gibt.
Der gezielte Proteinabbau (Targeted Protein Degradation, TPD) ist die populärste Methode für den Einsatz von proximitätsinduzierenden Substanzen. Sie wurde erstmals in den frühen 2000er Jahren demonstriert. Für die Entdeckung des Ubiquitin-vermittelten Proteinabbaus wurde sie 2004 mit dem Nobelpreis für Chemie ausgezeichnet. Das TPD-Konzept beinhaltet den selektiven Abbau von Proteinen durch das Hijacking interner zellulärer Maschinerien im menschlichen Körper. Im Vergleich zu besatzungsgesteuerten Methoden wie Inhibitoren haben TPD-Mechanismen den Vorteil, dass sie Gerüstfunktionen eliminieren und somit die Ursache des Problems angehen, anstatt nur Symptome zu behandeln. Um die biologische Homöostase aufrechtzuerhalten, werden im Allgemeinen Werkzeuge zur Beseitigung fehlgefalteter und denaturierter Proteine in einer Zelle benötigt.
Während verschiedene Mechanismen wie Lysosomen und Autophagie aktiv über PICs genutzt werden können, konzentriert sich Celeris Therapeutics auf das Ubiquitin-Proteasom-System. Auf der Grundlage dieses Systems werden unerwünschte Proteine mit Ubiquitin, einem kleinen Signalprotein, markiert. Dadurch wird der Abbau von pathogenen Proteinen eingeleitet.
Allerdings ist die Bewertung von TPD in einem experimentellen Laboraufbau langsam und teuer. Celeris Therapeutics hat daher einen computergestützten Workflow implementiert, um den Proteinabbau effektiv vorherzusagen und so die Zeitspanne der Medikamentenentwicklung zu verkürzen und zu rationalisieren.
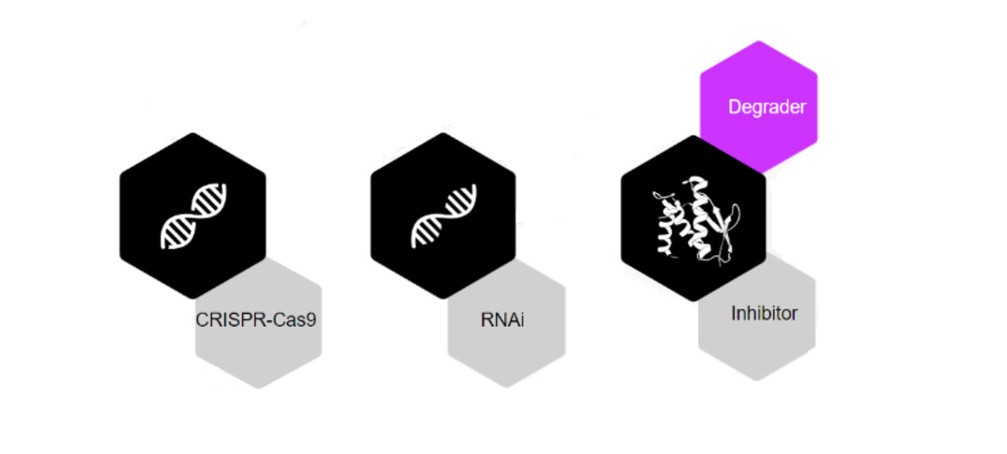
Proximitätsinduzierende Substanzen
Celeris Therapeutics (CelerisTx) leistet Pionierarbeit bei der Anwendung von KI auf PICs und konzentriert sich dabei auf den gezielten Proteinabbau. In Bezug auf Daten bedeutet das beim Proteinabbau, dass mehrere Moleküle identifiziert werden müssen, die auf eine bestimmte Weise miteinander interagieren. Stellen Sie sich LEGO®-Bausteine vor. Anstatt sie nur nach einer einzigen Dimension, z. B. der Form, zusammenzusetzen, haben diese Elemente mehrere Dimensionen oder Anforderungen zu erfüllen. Einige sind chemisch, biologisch, physikalisch angetrieben usw., was bedeutet, dass die Wechselwirkungen bestimmten Gesetzen unterliegen. Diese Gesetze müssen quantifiziert und in Daten eingebettet werden. Konkrete Beispiele für solche Informationen sind 3D-Strukturen von Molekülen, die als Graphen dargestellt werden, Hydrophobizität oder elektrostatisches Potenzial. Insgesamt handelt es sich um etwa 20 verschiedene Merkmale, die von Experten für TPD kuratiert werden. Infolgedessen werden enorme Datenmengen generiert, die mit Algorithmen des Machine Learning und insbesondere mit geometrischen Deep-Learning-Pipelines analysiert werden müssen. Eine konkrete ML-Anwendung ist erforderlich, um den Grad der Interaktion zwischen Molekülen zu bestimmen, die an einem sogenannten ternären Komplex beteiligt sind. Mit anderen Worten, wenn wir zwei Proteine betrachten, erwarten wir einen skalaren Wert, der den Grad der Interaktion bestimmt. Neben der Genauigkeit und Generalisierbarkeit müssen wir auch schnelle Vorhersagen in einem leistungsfähigen System treffen. Da wir verschiedene Protein-Protein-Wechselwirkungen simulieren, müssen wir ungenaue Vorhersagen schnell verwerfen, um den Bereich schnell zu analysieren. Dies hat sich bei einem Interaktionsdatensatz mit mehr als 20 000 Proteinpaaren als eine Herausforderung erwiesen.
Wie AWS für verschiedene PIC-Forschungsprojekte genutzt wurde
Um Entwicklerzeit zu sparen und einen schnelleren Einblick in die besonderen Probleme bei der Entwicklung von Machine Learning zu erhalten, haben wir uns an Amazon SageMaker gewandt. Dank dessen Funktionen konnten wir es vermeiden, einige Infrastrukturlösungen für Machine Learning selbst zu implementieren, z. B. die Erkennung von Abweichungen oder die Abstimmung der Hyperparameter. Die Erkennung von Abweichungen war bei der Datenaufbereitung von Bedeutung; daher nutzten wir SageMaker Clarify. Damit konnten wir die Qualität unserer Daten gewährleisten, noch bevor wir mit der Modellierung begannen. Sobald wir mit der Modellierung begonnen hatten, war es wichtig, direkte Integrationen von geometrischen Deep-Learning-Bibliotheken wie der SageMaker Deep Graph Library (DGL) zu haben. Da DGL ein Open-Source-Python-Paket für Deep Learning auf Diagrammen ist, nutzten wir es, um eine schnelle Einrichtung der für geometrisches Deep Learning erforderlichen Infrastruktur zu erhalten, was unerlässlich ist.
Sobald die anfänglichen Modelle festgelegt waren, verwendeten wir SageMaker Automatic Model Tuning, um schnell die besten Hyperparameter zu finden. Wir schätzen, dass wir dadurch Monate an Entwicklungszeit für die Programmierung der Hyperparameter-Optimierungs-Frameworks eingespart haben. Wir nutzten SageMaker Experiments, um alle Änderungen an den Experimenten zu verfolgen und zu organisieren. Diese Lösung war besonders wichtig. Bedenken Sie das bereits erwähnte Problem der Bestimmung der Interaktionsstärke zwischen den am ternären Komplex beteiligten Proteinen, die mehr als 15 geometrischen Deep-Learning-Architekturen, mit denen wir experimentiert haben, die Aufmerksamkeitsschichten, die reinen Faltungsoperatoren auf Diagrammen, usw. Die Nachvollziehbarkeit der verschiedenen Modelle und ihrer jeweiligen Parameter war wichtig, um festzustellen, was letztendlich funktionieren würde. Sobald wir die endgültige Version des Modells erstellt hatten, mussten wir sie debuggen und die Pipeline weiter optimieren. Hierfür erwies sich der SageMaker Debugger als hilfreich.
Zusammen mit den Softwarelösungen von SageMaker benötigten wir für unsere umfangreichen Daten- und Deep-Learning-Pipelines eine große Menge an Rechenleistung. Daher haben wir eine optimierte Spot-Flotten-Lösung für die GPUs entwickelt, die beim Training unserer Machine-Learning-Workflows zum Einsatz kommen.
Optimierung der Kosten unserer ML-Pipeline
Spot Instances sind eine besondere Art von Amazon-EC2-Instance, die 90 % Rabatt im Vergleich zu On-Demand-Preisen bieten. Manchmal kann es aufgrund einer hohen Nachfrage nach Rechenressourcen zu Unterbrechungen kommen, so dass ein Benutzer eine ganze Rechensitzung verliert. Es ist von entscheidender Bedeutung, die Zwischenergebnisse der Berechnung zu speichern, wenn man ein Deep-Learning-Modell über einen langen Zeitraum trainiert oder wenn eine zwangsläufig langfristige Berechnung auf der Spot Instance ausgeführt wurde. Abgesehen von der Speicherung der Zwischenergebnisse der Berechnung sollte der Benutzer auch in der Lage sein, die Berechnung automatisch auf einer anderen Spot Instance ab demselben Zwischenschritt fortzusetzen, bei dem die Verbindung unterbrochen wurde.
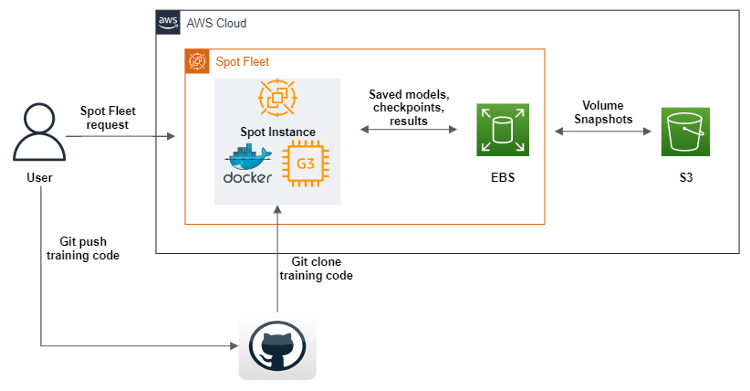
AWS SDK für Python (Boto3) wurde verwendet, um ein Skript zu erstellen, das bei der Ausführung eine Spot-Flotten-Anforderung sendet, um eine GPU-Spot-Instance mit Amazon Machine Images (AMI) zu erstellen, die die gesamte Software und Abhängigkeiten zur Ausführung unseres Machine-Learning-Codes enthält. Außerdem definieren wir Benutzerdatenskripts, die ein Docker-Image von Amazon Elastic Container Registery (Amazon ECR) herunterladen, und dann klont Git den Code aus unserem Repository. Schließlich führen die Benutzerdatenskripte den geometrischen Deep-Learning-Pipeline-Code aus. Dieser Code wurde so modifiziert, dass er nach jeder Trainingsepoche des Machine Learning Datensätze, Protokolle, Modelle und Checkpoints an ein Amazon-EBS-Volume sendet. Um sicherzustellen, dass die Objekte gespeichert werden, übertragen wir sie alle zu Amazon S3. Wir haben nicht nur eine Spot-Flotte definiert, sondern auch das Wiederauffüllen von Spot Instances aktiviert, die während des Prozesses getrennt werden. Dies ermöglicht es uns, das Training automatisch mit den letzten Checkpoints in den S3-Buckets fortzusetzen.
SageMaker bietet eine sehr ähnliche Lösung an, Managed Spot Training genannt. Wir haben das Angebot von SageMaker jedoch nicht genutzt, da die EC2-Instances, wenn sie als Teil von SageMaker verwendet werden, teurer sind als standardmäßige Instances. Das macht Sinn, denn SageMaker bietet viele ML-Angebote, ist einfach zu bedienen, erfordert weniger Entwicklungszeit usw. Für uns war die Entwicklung dieser Lösung eine strategische Zeitinvestition, denn wir wussten, dass wir GPUs über mehrere Jahre hinweg in verschiedenen Kapazitäten einsetzen würden.
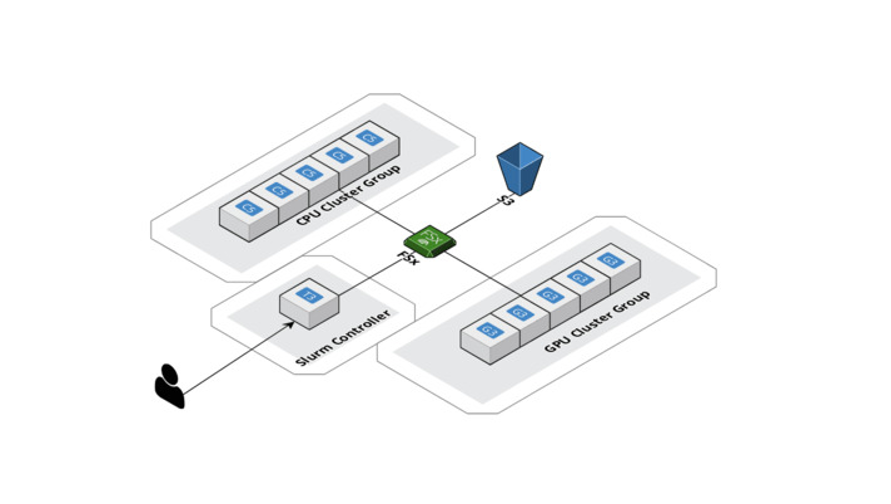
HPC war ein weiterer wichtiger Anwendungsfall, den wir mit AWS erfüllen mussten. Der riesige Datenraum, der mit geometrischem Deep Learning analysiert werden muss, ist mit einem erheblichen Rechenaufwand verbunden. Wir haben unsere Berechnungen mit AWS ParallelCluster horizontal hochskaliert und ein gemeinsam genutztes Ablagesystem, Amazon FSx für Lustre, erstellt, so dass alle Rechenknoten von der gleichen Stelle aus auf die Daten zugreifen und sie ändern konnten. Außerdem unterstützt AWS Slurm als Workload-Manager, der es uns ermöglicht, die Arbeit auf CPUs und GPUs gleichzeitig zu verteilen. Dies ermöglicht letztendlich nicht nur schnellere Experimente, sondern auch eine optimierte Modellbereitstellung. Es sei noch einmal darauf hingewiesen, dass SageMaker einen ähnlichen Service für große Datensätze und Modelle anbietet, die so genannten Distributed Training Libraries.
Die Zukunft für proximitätsinduzierende Substanzen
Vorläufige Ergebnisse aus unserer Pipeline liegen für die Parkinson-Krankheit vor, die in vorklinische Studien übergeht. Schnelles und zuverlässiges Experimentieren ist für die computergestützte Arzneimittelforschung von entscheidender Bedeutung, da wir unsere Arzneimittelforschungspipeline mit neuen Architekturen und Ansätzen weiter verbessern. Unser F&E-Ansatz, bei dem wir beträchtliche Ressourcen in die Forschung investieren, bedeutet, dass wir mit vielen laufenden Risiken konfrontiert sind, die wir abfedern müssen. Die Standardangebote von AWS sind eine Möglichkeit, um sicherzustellen, dass wir agil sind und schneller von der Forschung ins Labor und dann auf den Markt gelangen.
Fazit
Jetzt ist es an der Zeit, die Produktivität in der Arzneimittelforschung zu steigern. Die Umkehrung des Mooreschen Gesetzes in der Pharmakologie, bekannt als Eroom‘s Law, zeigt, dass die Forschungs- und Entwicklungskosten für die Entwicklung eines neuen Medikaments weiter steigen werden. Ein Ansatz zur Lösung dieses Problems besteht darin, den Prozess kontinuierlicher Machine-Learning-Experimente für die Arzneimittelforschung zu nutzen und zu optimieren.

Christopher Trummer
Christopher Trummer ist Mitgründer von Celeris Therapeutics und fungiert als CEO. Er war mehrfach eingeladener Hauptredner im Rahmen von Konferenzen über KI für die Wirkstoffforschung und ist Mitautor von von Experten begutachteten Veröffentlichungen in verschiedenen Zeitschriften.

Noah Weber
Noah Weber ist Chief Technology Officer bei Celeris Therapeutics. Er ist Kaggle Grandmaster und Lehrbeauftragter an der Technischen Universität Wien und der Fachhochschule Wien.

Olajide Enigbokan
Olajide Enigbokan ist Startup Solutions Architect bei Amazon Web Services. Er liebt es, mit Startups (insbesondere mit Entwicklern) zusammenzuarbeiten, um ihnen den Wert der AWS Cloud zu erläutern.

AWS Editorial Team
Das Content Marketing Team von AWS Startups arbeitet mit Startups aller Größen und Branchen zusammen, um außergewöhnliche Inhalte bereitzustellen, die informieren, unterhalten und inspirieren.
Wie war dieser Inhalt?