Como estava esse conteúdo?
- Aprenda
- CelerisTx: descoberta de medicamentos para doenças incuráveis com ML na AWS
CelerisTx: descoberta de medicamentos para doenças incuráveis com ML na AWS
Aproveitando a descoberta computacional de medicamentos em compostos indutores de proximidade (PICs)
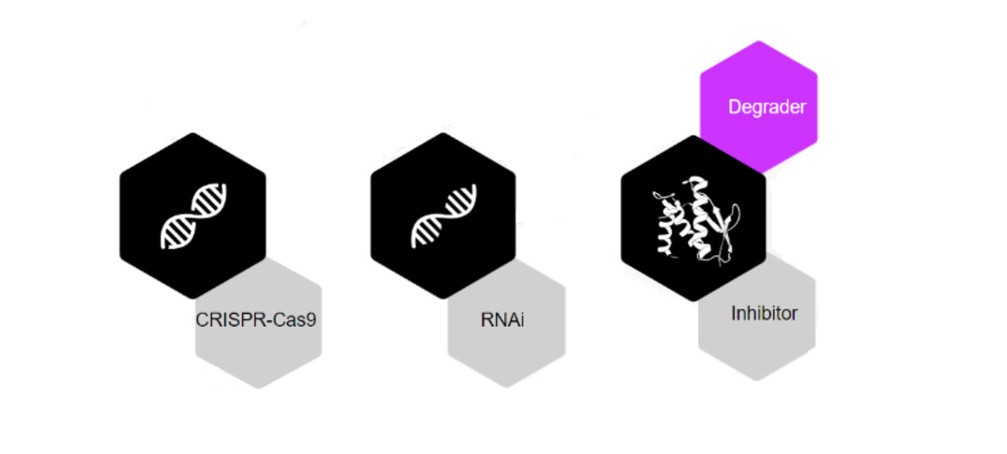
A humanidade avançou com inúmeras soluções terapêuticas para tratar doenças. Tecnologias recentes, como nocaute (CRISPR — funciona no nível do DNA) e nocaute (RNAi — funciona no nível do RNA), têm limitações significativas. Em contraste, os compostos indutores de proximidade (PICs) combinam as vantagens exclusivas de moléculas pequenas. Essas características benéficas incluem biodisponibilidade oral e facilidade de fabricação — junto com as de tecnologias de silenciamento, como CRISPR e RNAi. Em contraste com o RNAi e o CRISPR, os PICs afetam as proteínas, fornecendo assim uma modalidade terapêutica inovadora para direcionar proteínas, especificamente associadas a doenças. As alternativas que têm como alvo as proteínas incluem produtos biológicos e inibidores, e essas alternativas não expandiram a gama de proteínas patogênicas que podem ser direcionadas. Mais de 80% das proteínas patogênicas estão associadas a uma doença ainda não passível de intervenções farmacêuticas.
A degradação proteica direcionada (TPD) é a modalidade de fármaco indutora de proximidade mais popular, demonstrada pela primeira vez no início dos anos 2000. Também recebeu o Prêmio Nobel de Química por descobrir a degradação de proteínas mediada por ubiquitina em 2004. O conceito de TPD envolve a degradação seletiva de proteínas por meio do sequestro da maquinaria celular interna do corpo humano. Em comparação com métodos orientados pela ocupação, como inibidores, os mecanismos de TPD têm a vantagem de eliminar as funções de andaime e, assim, abordar a causa raiz do problema, em vez de apenas tratar os sintomas. Em geral, ferramentas para eliminar proteínas mal dobradas e desnaturadas dentro de uma célula são necessárias para manter a homeostase biológica.
Embora várias máquinas possam ser utilizadas ativamente por meio de PICs, como lisossomos e autofagia, a Celeris Therapeutics se concentra no sistema ubiquitina-proteassoma. Com base nesse sistema, proteínas indesejadas são marcadas com ubiquitina, uma pequena proteína sinalizadora. Como resultado, esse processo inicia a degradação de proteínas patogênicas.
No entanto, a avaliação do TPD em uma configuração de laboratório experimental é lenta e cara. Portanto, a Celeris Therapeutics implementou um fluxo de trabalho computacional para prever a degradação de proteínas de forma eficaz e, assim, encurtar e agilizar o cronograma de desenvolvimento de medicamentos.
Compostos indutores de proximidade
A Celeris Therapeutics (CelerisTx) é pioneira na adoção de IA em PICs, com foco na degradação direcionada de proteínas. Em termos de dados, na degradação de proteínas, isso significa identificar várias moléculas que interagem umas com as outras de determinada maneira. Imagine peças LEGO®. Em vez de encaixá-las de acordo com uma única dimensão ou forma, esses elementos têm várias dimensões ou requisitos para satisfazer. Alguns são químicos, biológicos, físicos etc., o que significa que as interações estão sujeitas a leis específicas. Essas leis precisam ser quantificadas e incorporadas aos dados. Exemplos concretos de tais informações incluem estruturas 3D de moléculas representadas como gráficos, hidrofobicidade ou potencial eletrostático. No total, isso equivale a cerca de 20 recursos diferentes que os especialistas em TPD selecionam. Como resultado, uma enorme quantidade de dados está sendo gerada e precisa ser analisada usando algoritmos de machine learning e, especificamente, pipelines geométricos de aprendizado profundo. Uma aplicação concreta de ML é necessária para determinar o grau de interação entre as moléculas envolvidas no chamado complexo ternário. Em outras palavras, considerando duas proteínas, esperamos um valor escalar que determine o grau de interação. Além de sermos precisos e generalizáveis, precisamos fazer previsões rápidas em um sistema de alto performance. À medida que simulamos diferentes interações proteína-proteína, é necessário descartar previsões imprecisas rapidamente para analisá-las imediatamente no espaço. Isso provou ser um desafio para um conjunto de dados de interação com mais de 20 mil pares de proteínas.
Como a AWS foi aproveitada para vários projetos de descoberta de PIC
Para economizar tempo do desenvolvedor e obter uma visão mais rápida dos problemas específicos relacionados ao desenvolvimento de machine learning, recorremos ao Amazon SageMaker. Seus recursos nos permitiram evitar a implementação de algumas soluções de infraestrutura de machine learning sozinhos, como detecção de viés ou ajuste de hiperparâmetros. A detecção de viés foi relevante durante a preparação dos dados; portanto, utilizamos o SageMaker Clarify. Isso foi fundamental para garantir a qualidade de nossos dados, antes mesmo de começarmos a modelar. Posteriormente, depois de começarmos a modelar, era importante ter integrações diretas de bibliotecas geométricas de aprendizado profundo, como a SageMaker Deep Graph Library (DGL). Como o DGL é um pacote python de código aberto para aprendizado profundo em grafos, nós o usamos para obter uma configuração rápida da infraestrutura necessária para o aprendizado profundo geométrico, o que é essencial.
Depois que os modelos iniciais foram definidos, para encontrar os melhores hiperparâmetros rapidamente, usamos o SageMaker Automatic Model Tuning. Estimamos que, assim, evitamos meses de tempo de desenvolvimento na codificação das estruturas de otimização de hiperparâmetros. Aproveitamos o SageMaker Experiments para rastrear e organizar todas as mudanças nos experimentos. Essa solução foi especialmente importante. Considere o problema acima mencionado de determinar a força da interação entre proteínas envolvidas no complexo ternário, as mais de 15 arquiteturas geométricas de aprendizado profundo que experimentamos a partir de camadas de atenção, operadores de convolução pura em gráficos, etc. A rastreabilidade dos diferentes modelos e seus respectivos parâmetros foi importante para determinar o que eventualmente funcionaria. Depois de criarmos a versão final do modelo, precisávamos depurá-los e otimizar ainda mais o pipeline. Para isso, o SageMaker Debugger se mostrou útil.
Junto com as soluções de software do SageMaker, precisávamos de muito poder de computação para nossos vastos pipelines de dados e aprendizado profundo. Por isso, criamos uma solução Spot Fleet otimizada para as GPUs usadas no treinamento de nossos fluxos de trabalho de machine learning.
Otimizando o custo do nosso pipeline de ML
As instâncias spot são um tipo especial de instância do Amazon EC2 que oferecem 90% de desconto em comparação aos preços sob demanda. Às vezes, devido à alta demanda por recursos de computação, os recursos de computação podem ser interrompidos, fazendo com que o usuário perca uma sessão de computação inteira. É crucial salvar os resultados intermediários da computação, se alguém treinar um modelo de aprendizado profundo por um longo tempo ou se alguma computação necessariamente de longa duração foi executada na Instância Spot. Além de salvar os resultados intermediários da computação, o usuário também deve poder continuar automaticamente a computação em outra instância spot a partir da mesma etapa intermediária em que a desconexão ocorreu.
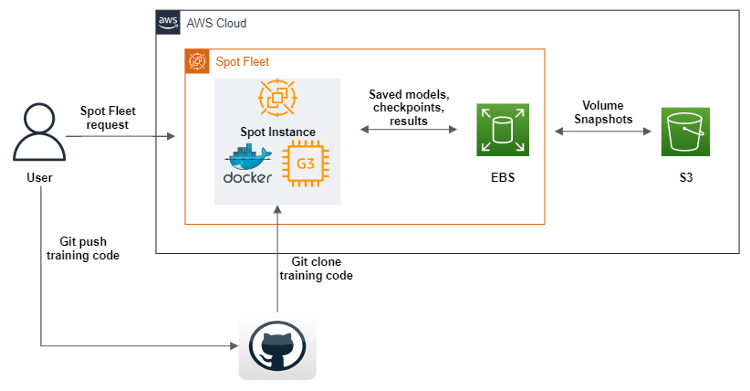
O AWS SDK para Python (boto3) foi usado para criar um script que, após a execução, enviará uma solicitação Spot Fleet para criar uma instância spot de GPU com imagens de máquina da Amazon (AMIs), contendo todo o software e dependências para executar nosso código de machine learning. Além disso, definimos scripts de dados do usuário que baixam uma imagem do Docker do Amazon Elastic Container Registery (Amazon ECR) e, em seguida, o Git clona o código do nosso repositório. Finalmente, os scripts de dados do usuário executam o código geométrico do pipeline de aprendizado profundo. Esse código foi modificado para que, após cada período de treinamento de machine learning, ele envie conjuntos de dados, registros, modelos e pontos de verificação para um volume do Amazon EBS. Para garantir que os objetos sejam salvos, despejamos todos eles no Amazon S3. Além de definir uma frota spot, também ativamos o reabastecimento de qualquer instância spot que se desconecte no processo, o que nos permite continuar o treinamento automaticamente a partir dos últimos pontos de verificação encontrados nos buckets do S3.
O SageMaker oferece algo muito semelhante, chamado Managed Spot Training. No entanto, não aproveitamos a oferta do SageMaker porque, quando usadas como parte do SageMaker, as instâncias do EC2 são mais caras do que as padrão. Isso faz sentido, já que o SageMaker oferece muitas ofertas de ML, facilidade de uso, menos tempo de engenharia necessário, etc. Para nós, arquitetar essa solução foi um investimento estratégico de tempo, porque sabíamos que usaríamos GPUs em diferentes capacidades por vários anos.
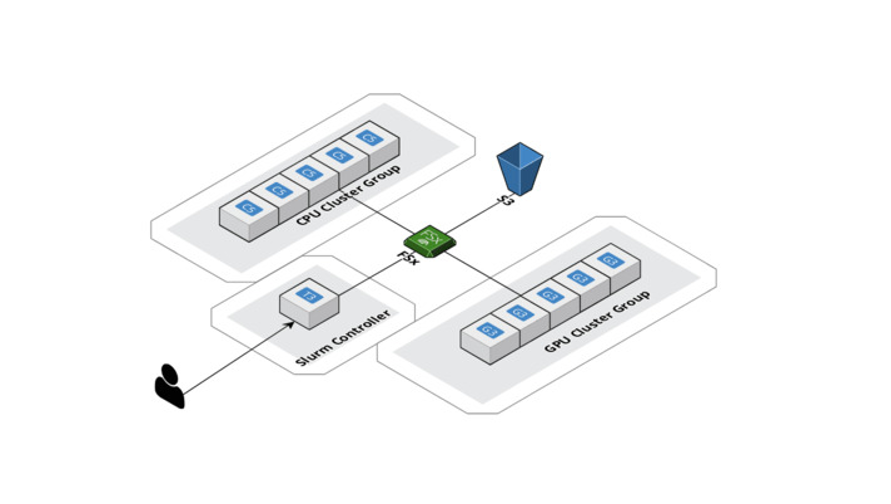
A HPC foi outro caso de uso significativo que precisávamos cumprir com a AWS. O enorme espaço de dados que precisa ser analisado com o aprendizado profundo geométrico vem com uma carga computacional substancial. Aumentamos a escala dos nossos cálculos horizontalmente com o AWS ParallelCluster e criamos um sistema de arquivos compartilhado, o Amazon FSx para Lustre, para que todos os nós de computação pudessem acessar e modificar dados do mesmo local. Além disso, a AWS oferece suporte ao Slurm como gerenciador de workload, o que nos permite distribuir o trabalho entre CPUs e GPUs ao mesmo tempo. Eventualmente, isso permite não apenas uma experimentação mais rápida, mas também uma exibição otimizada do modelo. Novamente, deve-se observar que o SageMaker oferece um serviço similar para grandes conjuntos de dados e modelos, chamado de Bibliotecas de Treinamento Distribuídas.
O futuro dos compostos indutores de proximidade
Os resultados preliminares de nosso pipeline estão disponíveis para a doença de Parkinson que evolui para estudos pré-clínicos. Experimentar de forma rápida e confiável é essencial para a descoberta computacional de medicamentos, à medida que continuamos aprimorando nosso pipeline de descoberta de medicamentos com novas arquiteturas e abordagens. Nossa abordagem de P&D, na qual investimos recursos substanciais em pesquisa, significa que enfrentamos muitos riscos contínuos que precisamos mitigar. As ofertas padrão da AWS são uma forma de garantir que sejamos ágeis e passemos mais rapidamente da pesquisa para o laboratório e, em seguida, para o mercado.
Conclusão
Agora é a hora de aumentar a produtividade na descoberta de medicamentos. O inverso da Lei de Moore em farmacologia, conhecida como “Lei de Eroom”, indica que os custos de pesquisa e desenvolvimento necessários para desenvolver um novo medicamento só continuarão aumentando. Uma abordagem para resolver isso é aproveitar e agilizar o processo de realização de experimentos sustentados de machine learning para a descoberta de medicamentos.

Christopher Trummer
Christopher Trummer é cofundador da Celeris Therapeutics e atua como CEO. Ele foi diversas vezes palestrante convidado em Inteligência Artificial de conferências sobre a descoberta de medicamentos e é coautor de publicações resenhadas por seus pares em vários periódicos.

Noah Weber
Noah Weber atua como Chefe de Tecnologia da Celeris Therapeutics. Ele é Grão-Mestre da Kaggle e professor adjunto na Universidade de Tecnologia de Viena e na Universidade de Ciências Aplicadas de Viena.

Olajide Enigbokan
Olajide Enigbokan é Arquiteto de Soluções de Startups na Amazon Web Services. Ele adora trabalhar com startups (principalmente desenvolvedores) para que descubram o valor da Nuvem AWS.

AWS Editorial Team
A equipe de Marketing de Conteúdo da AWS Startups colabora com startups de todos os tamanhos e setores para oferecer excepcional conteúdo educativo, divertido e inspirador.
Como estava esse conteúdo?