Comment a été ce contenu ?
- Apprendre
- CelerisTx : découverte de médicaments pour les maladies incurables avec le ML sur AWS
CelerisTx : découverte de médicaments pour les maladies incurables avec le ML sur AWS
Exploiter la découverte informatique de médicaments sur des composés induisant la proximité (PIC)
L'humanité a évolué avec d'innombrables solutions thérapeutiques pour traiter les maladies. Les technologies récentes telles que le knock-out (CRISPR au niveau de l'ADN) et le knock-down (ARNi au niveau de l'ARN) présentent des limites importantes. En revanche, les composés induisant la proximité (PIC) combinent les avantages uniques des petites molécules. Ces fonctionnalités bénéfiques comprennent la biodisponibilité orale et la facilité de fabrication, ainsi que celles des technologies d'inhibition telles que CRISPR et l'ARNi. Contrairement à l'ARNi et à CRISPR, les PIC affectent les protéines, ce qui constitue une modalité thérapeutique révolutionnaire pour cibler les protéines spécifiquement associées aux maladies. Les alternatives qui ciblent les protéines comprennent les produits biologiques et les inhibiteurs, et ces alternatives n'ont pas élargi la gamme des protéines pathogènes qui peuvent être ciblées. Plus de 80 % des protéines pathogènes sont associées à une maladie qui ne peut pas encore faire l'objet d'interventions pharmaceutiques.
La dégradation ciblée des protéines (TPD) est la modalité médicamenteuse induisant la proximité la plus populaire, démontrée pour la première fois au début des années 2000. Elle a également reçu un prix Nobel de chimie pour avoir découvert la dégradation des protéines induite par l'ubiquitine en 2004. Le concept de TPD implique la dégradation sélective des protéines en détournant la machinerie cellulaire interne du corps humain. Par rapport aux méthodes axées sur l'occupation telles que les inhibiteurs, les mécanismes de la TPD ont l'avantage d'éliminer les fonctions d'échafaudage et de s'attaquer ainsi à la cause première du problème plutôt que de simplement traiter les symptômes. En général, des outils permettant d'éliminer les protéines mal repliées et dénaturées au sein d'une cellule sont nécessaires pour maintenir l'homéostasie biologique.
Alors que divers mécanismes tels que les lysosomes et l'autophagie peuvent être activement utilisés à travers les PIC, Celeris Therapeutics se concentre sur le système ubiquitine-protéasome. Sur la base de ce système, les protéines indésirables sont marquées avec de l'ubiquitine, une petite protéine de signalisation. Ce processus initie donc la dégradation de protéines pathogènes.
Cependant, l'évaluation de la TPD dans un laboratoire expérimental est lente et coûteuse. Celeris Therapeutics a donc mis en œuvre un flux de travail informatique pour prévoir efficacement la dégradation des protéines et ainsi raccourcir et rationaliser le calendrier de développement des médicaments.
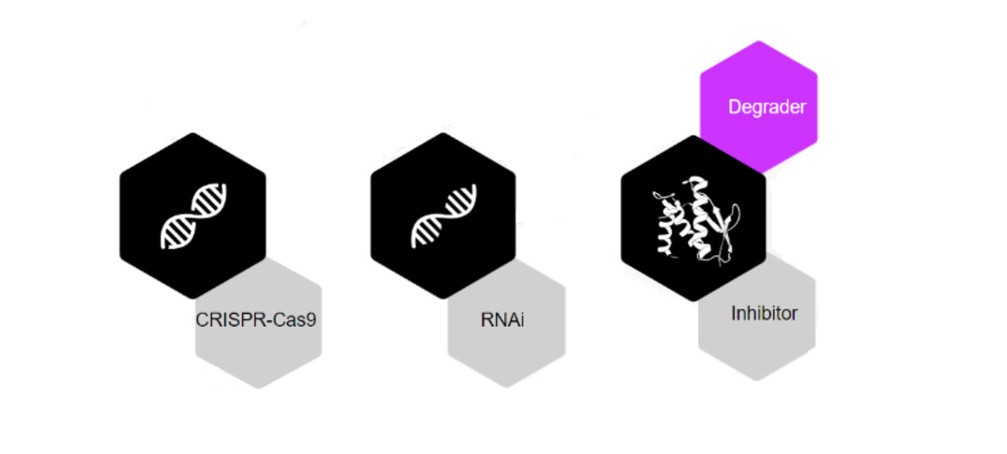
Composés induisant la proximité
Celeris Therapeutics (CelerisTx) est la première entreprise à adopter l'IA pour les PIC, en se concentrant sur la dégradation ciblée des protéines. En termes de données, dans la dégradation des protéines, il s'agit d'identifier plusieurs molécules qui interagissent les unes avec les autres d'une manière particulière. Imaginez des briques LEGO®. Au lieu de les assembler en fonction d'une seule dimension, à savoir la forme, ces éléments doivent répondre à de multiples dimensions ou exigences. Certains sont chimiques, biologiques, physiques, etc., ce qui signifie que les interactions sont soumises à des lois spécifiques. Ces lois doivent être quantifiées et intégrées dans des données. Parmi les exemples concrets de ces informations, on peut citer les structures 3D des molécules représentées sous forme de graphiques, l'hydrophobie ou le potentiel électrostatique. Au total, ce sont environ 20 fonctionnalités différentes que les experts de la TPD conservent. Par conséquent, une énorme quantité de données est générée et doit être analysée à l'aide d'algorithmes de machine learning, et plus particulièrement, de pipelines de deep learning géométrique. Une application concrète de ML est nécessaire pour déterminer le degré d'interaction entre les molécules impliquées dans un complexe ternaire. En d'autres termes, en considérant deux protéines, nous attendons une valeur scalaire qui détermine le degré d'interaction. En plus d'être précis et généralisable, nous devons faire des prédictions rapides dans un système performant. Comme nous simulons différentes interactions protéine-protéine, il est nécessaire d'écarter rapidement les prédictions inexactes afin d'analyser rapidement l'espace. Cette démarche s'est avérée difficile pour un jeu de données d'interactions comprenant plus de 20 000 paires de protéines.
Comment AWS a été utilisé pour divers projets de découverte de PIC
Pour faire gagner du temps aux développeurs et mieux comprendre les problèmes spécifiques liés au développement du machine learning, nous nous sommes tournés vers Amazon SageMaker. Ses fonctionnalités nous ont permis d'éviter de mettre en œuvre nous-mêmes certaines solutions d'infrastructure de machine learning comme la détection de biais ou le réglage des hyperparamètres. La détection des biais était pertinente lors de la préparation des données. C'est pourquoi nous avons tiré parti de SageMaker Clarify. Nous avons ainsi pu garantir la qualité de nos données, avant même que nous ne commencions à modéliser. Ensuite, une fois que nous avons commencé à modéliser, il était important d'intégrer directement les bibliothèques géométriques de deep learning, notamment la bibliothèque SageMaker Deep Graph (DGL). DGL étant un package Python open source pour le deep learning sur les graphes, nous l'avons utilisé pour la configuration rapide de l'infrastructure nécessaire au deep learning géométrique, ce qui est essentiel.
Une fois les modèles initiaux définis, nous avons utilisé SageMaker Automatic Model Tuning pour trouver rapidement les meilleurs hyperparamètres. Nous estimons avoir ainsi gagné des mois de développement lors du codage des cadres d'optimisation des hyperparamètres. Nous avons tiré parti des expériences SageMaker pour suivre et organiser toutes les modifications apportées aux expériences. Cette solution était particulièrement importante. Examinons le problème susmentionné qui consiste à déterminer la force de l'interaction entre les protéines impliquées dans le complexe ternaire, les plus de 15 architectures géométriques de deep learning que nous avons expérimentées à partir de couches d'attention, les opérateurs de convolution purs sur les graphes, etc. La traçabilité des différents modèles et de leurs paramètres respectifs était importante pour déterminer ce qui fonctionnerait finalement. Une fois que nous avons créé la version finale du modèle, nous avons dû les déboguer et optimiser davantage le pipeline. À cet égard, SageMaker Debugger s'est révélé utile.
Outre les solutions logicielles de SageMaker, nous avions besoin d'une puissance de calcul importante pour nos vastes pipelines de données et de deep learning. C'est pourquoi nous avons développé une solution de parc d'instances Spot optimisée pour les GPU utilisés dans la formation de nos flux de travail de machine learning.
Optimisation du coût de notre pipeline de machine learning
Les instances Spot sont un type spécial d'instance Amazon EC2 qui offre une réduction de 90 % par rapport aux prix à la demande. Parfois, en raison de la forte demande de ressources informatiques, celles-ci peuvent être interrompues, entraînant la perte d'une session de calcul complète par l'utilisateur. Il est crucial de sauvegarder les résultats intermédiaires du calcul, si l'on entraîne un modèle de deep learning pendant une longue période ou si des calculs nécessairement longs ont été exécutés sur l'instance Spot. Outre la sauvegarde des résultats intermédiaires du calcul, l'utilisateur doit également être en mesure de poursuivre automatiquement le calcul sur une autre instance Spot à partir de la même étape intermédiaire que celle où la déconnexion s'est produite.
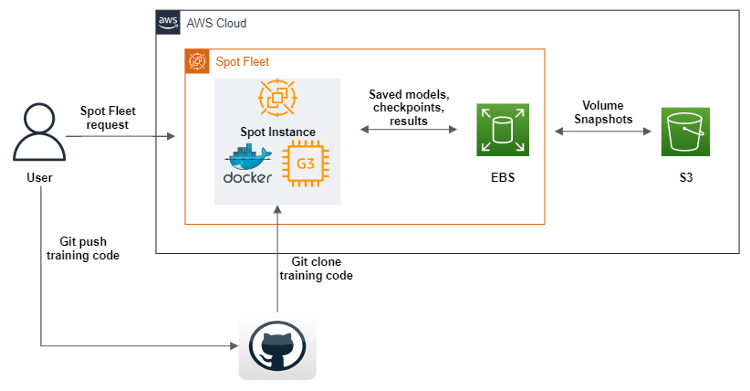
Le kit AWS SDK pour Python (boto3) a été utilisé pour créer un script qui, lors de son exécution, enverra une requête de parc d'instances Spot pour créer une instance Spot GPU avec Amazon Machine Images (AMI) contenant tous les logiciels et dépendances nécessaires à l'exécution de notre code de machine learning. Nous définissons également des scripts de données utilisateur qui téléchargent une image docker depuis Amazon Elastic Container Registery (Amazon ECR), puis Git clone le code depuis notre référentiel. Enfin, les scripts de données utilisateur exécutent le code géométrique du pipeline de deep learning. Ce code a été modifié de telle sorte qu'après chaque période d'entraînement au machine learning, il envoie des jeux de données, des journaux, des modèles et des points de contrôle à un volume Amazon EBS. Pour nous assurer que les objets sont enregistrés, nous les déposons tous sur Amazon S3. Nous avons non seulement défini un parc d'instances Spot, mais aussi activé le réapprovisionnement de toutes les instances Spot qui se déconnectent au cours du processus, ce qui nous permet de poursuivre l'entraînement automatiquement à partir des derniers points de contrôle trouvés dans les compartiments S3.
SageMaker propose quelque chose de très similaire, appelé Managed Spot Training. Cependant, nous n'avons pas tiré parti de l'offre SageMaker, car lorsqu'elles sont utilisées dans le cadre de SageMaker, les instances EC2 sont plus chères que les instances standard. Cela est tout à fait logique, car SageMaker propose de nombreuses offres de machine learning, une facilité d'utilisation, une réduction du temps d'ingénierie, entre autres. Pour nous, l'architecture de cette solution représentait un investissement de temps stratégique, car nous savions que nous allions utiliser des GPU à différentes capacités pendant plusieurs années.
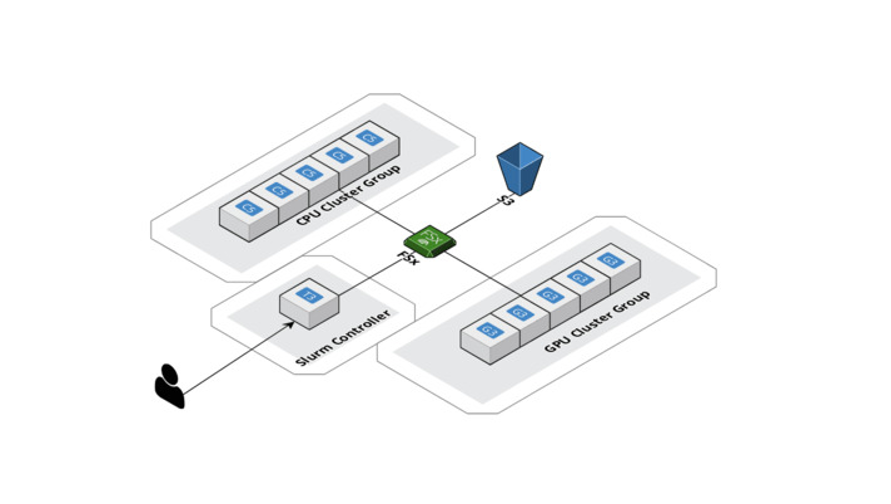
Le HPC était un autre cas d'utilisation important que nous devions résoudre avec AWS. L'énorme espace de données qui doit être analysé à l'aide du deep learning géométrique entraîne une charge de calcul importante. Nous avons augmenté nos calculs avec AWS ParallelCluster et créé un système de fichiers partagé, Amazon FSx pour Lustre, afin que tous les nœuds de calcul puissent accéder aux données et les modifier depuis le même endroit. Par ailleurs, AWS prend en charge Slurm en tant que gestionnaire de charges de travail, ce qui nous permet de répartir le travail entre les processeurs et les GPU en même temps. Nous pouvons ainsi à terme non seulement accélérer l'expérimentation, mais également optimiser la diffusion des modèles. Encore une fois, il convient de noter que SageMaker propose un service similaire pour les grands jeux de données et modèles, appelé bibliothèques de formation distribuées.
L'avenir des composés induisant la proximité
Les résultats préliminaires de notre pipeline sont disponibles pour la maladie de Parkinson qui progresse vers des études précliniques. L'expérimentation rapide et fiable est essentielle pour la découverte informatisée de médicaments, car nous continuons à améliorer notre pipeline de découverte de médicaments avec de nouvelles architectures et approches. Notre approche en matière de recherche et de développement, dans le cadre de laquelle nous investissons des ressources substantielles dans la recherche, implique de nombreux risques permanents que nous devons atténuer. Les offres standard d'AWS sont un moyen de garantir notre agilité et de passer plus rapidement de la recherche au laboratoire, puis au marché.
Conclusion
Le moment est venu d'accroître la productivité dans le domaine de la découverte de médicaments. En pharmacologie, l'inverse de la loi de Moore, connue sous le nom de « loi d'Eroom », indique que les coûts de recherche et de développement nécessaires à la mise au point d'un nouveau médicament ne cesseront d'augmenter. Une approche pour y remédier consiste à exploiter et à rationaliser le processus de réalisation d'expériences soutenues de machine learning pour la découverte de médicaments.

Christopher Trummer
Christopher Trummer est co-fondateur de Celeris Therapeutics et en est le PDG. Il a été à de nombreuses reprises invité en tant que conférencier (dans le domaine de l’IA) lors de conférences sur la découverte de médicaments. Il est également co-auteur de publications évaluées par des pairs dans diverses revues.

Noah Weber
Noah Weber est directeur technique chez Celeris Therapeutics. Il est grand maître Kaggle et professeur adjoint à l'université technique et à l'université des sciences appliquées de Vienne.

Olajide Enigbokan
Olajide Enigbokan est architecte de solutions pour les startups Amazon Web Services. Il adore travailler avec des startups (en particulier celles spécialisées dans le domaine de la construction) et découvrir la valeur du cloud AWS.

AWS Editorial Team
L'équipe de marketing de contenu d'AWS Startups collabore avec des startups de toutes tailles et de tous secteurs pour proposer un contenu exceptionnel qui éduque, divertit et inspire.
Comment a été ce contenu ?