Amazon Web Services ブログ
Part 2: Amazon Omics を使用したゲノミクスデータの保管と分析のエンドツーエンド自動化
AWS のソリューションアーキテクトである David Obenshain 氏、Gargi Singh Chhatwal 氏、Joshua Broyde 氏の価値ある貢献に感謝します。
研究機関や医療機関においてのゲノムシーケンシングの急速な進歩と活用に伴い、データとコンピューティングリソースを管理するための、信頼性が高く、再現性があり、スケーラブルで、コスト効率の高いインフラストラクチャが必要になっています。お客様はインフラストラクチャのセットアップと保守という差別化されない重労働に費やす時間を削減し、代わりに科学的なビジネス上の差別化要因に集中したいと考えています。
このブログ記事の Part 1 では、ヘルスケアおよびライフサイエンスのお客様が直面するゲノムデータやその他の生物学的データの保存と処理に関するビジネス上の課題について説明しました。これらの課題に具体的に対処するための専用サービスとして Amazon Omics を紹介しました。Amazon Omics はバイオインフォマティシャン、研究者、科学者を支援します。
・ソースデータおよび加工済データの保存
・バイオインフォマティクスパイプラインを通じたソースデータの処理
・サンプルのコホート全体で洞察を生成するためのゲノムバリアントのクエリと分析
このブログ記事では、シーケンスデータをインサイトにシームレスに変換するプロセス自動化のために、Amazon Omics と AWS Step Functions をどのように使用するかご紹介します。入力された生のシーケンスデータの保存と分析を、クエリ可能なバリアントに至るまで自動化するサンプルコードを用いたリファレンスアーキテクチャの例を説明します。このサンプルコードはデモンストレーション用であり、実稼働用ではないことに注意してください。
ソリューションの概要
このソリューションは、きめ細かなアクセス許可、Amazon Simple Storage Service (Amazon S3) のライフサイクルポリシー、AWS CloudFormation を使用した Infrastructure as Code などのベストプラクティスに基づいて構築されています。利用者は必要に応じてこれらをカスタマイズすることができます。
このソリューションはサーバレスサービスを使用し、手動による設定を最小限に抑えるように設計されているため、お客様の運用負担を削減することができます。AWS Step Functions と AWS Lambda (Lambda) とのネイティブ統合を使用して、Amazon Omics によるゲノミクスデータの保存、分析、クエリに必要なタスクのオーケストレーションを自動化しています。
ゲノムデータをバリアントに変換するために Broad Institute の GATK ベストプラクティスをベースにしたサンプルワークフローを使用しました。また、このワークフローに必要な GATK Resource Bundle の公開データも使用しています。
このソリューションの一部である AWS CloudFormation テンプレートは、一度デプロイすると、次の AWS リソースを作成します。
- シーケンスインプットデータとワークフローのアウトプットデータ用のライフサイクルルールを含む S3 バケット。これによりデータは Amazon Omics のストレージにロードされた後、低コストのストレージクラスに移行されるか削除されます。
- 自動化に必要な Lambda 関数と Docker イメージ (アカウントのプライベート Amazon Elastic Container Registry にプッシュされる) を構築およびデプロイする AWS Code Build。
- データストレージ、二次解析ワークフロー、バリアントデータの取り込みに必要な Amazon Omics リソース:
- オミックスリファレンスストアおよびリファレンスゲノムの自動インポート
- オミックスシーケンスストア
- オミックスワークフローとサンプルの二次分析ワークフローの自動生成
- オミックスバリアントストア (
omicsvariantstore) - ClinVar VCF の自動インポート機能を備えたオミックスアノテーションストア (
omicsannotationstore)
- オミックスシーケンスストアとオミックスバリアントストアへのデータインポートおよび事前作成されたオミックスワークフローの自動起動をオーケストレートする Step Function ワークフロー。
- 様々なリソースの作成と起動をトリガーする Lambda 関数と Amazon S3 通知。
 図 1: Amazon Omics、AWS Step Functions、AWS Lambda を使用してゲノミクスデータを保存および分析する自動化されたエンドツーエンドソリューションのリファレンスアーキテクチャ
図 1: Amazon Omics、AWS Step Functions、AWS Lambda を使用してゲノミクスデータを保存および分析する自動化されたエンドツーエンドソリューションのリファレンスアーキテクチャ
リソースが作成されると、FASTQ 形式のゲノム入力データを処理する準備が整います。図 1 はデータが処理される際の一連のイベントを示しています。
- ユーザはインプット用に指定された既存 S3 バケットに FASTQ 形式のシーケンスデータをアップロードします。
- インプット用 S3 バケットには Lambda 関数をトリガーするように設定された Amazon S3 イベント通知が設定されています。Lambda 関数はファイル名を評価し、同じサンプル名の FASTQ ファイルのペアが検出されると AWS Step Functions ワークフローをトリガーします。
- AWS Step Functions ワークフローには以下 Lambda 関数を呼び出すステップが含まれています。
- FASTQ ファイルを既存のオミックスシーケンスストアにインポートします。
- 二次解析のための GATK ベストプラクティスオミックスワークフローを使用する、作成済オミックスワークフローを開始します。
- ワークフローが完了したら、ワークフローのアウトプットの BAM と VCF をオミックスシーケンスストアとオミックスバリアントストアにそれぞれインポートします。
- Amazon S3 のインプットファイルとアウトプットファイルにタグをつけて、ライフサイクル設定に従ってよりコールドなストレージクラスに移動するか、時間の経過とともに削除されるようにします。
- バリアントストアとアノテーションストアのテーブルは AWS Lake Formationのリソースとして表示されます。
バリアントデータのクエリ
オミックスバリアントストアおよびアノテーションストアにインポートされたバリアントとアノテーションデータは Amazon Omics と AWS Lake Formation (Lake Formation) の統合を使用して共有テーブルとして表示されます。これによりユーザーはきめ細やかなアクセス制御を実装できます。上記のサンプルコードを使用すると、omicsvariantstoreテーブルと omicsannotationstoreテーブルが Lake Formation に表示されます。
Lake Formation により、データレイク管理者は AWS Identity and Access Management (IAM) のユーザーとロールに目的のデータベースとテーブルへのアクセス権を付与できます。特定のユーザーがデータをクエリするための適切なレベルの権限をもつために管理者が必要となる手順について以下説明します。権限が付与されると、ユーザーは Amazon Athena (Athena) を使用してバリアントデータとアノテーションデータにアクセスできます。
AWS Lake Formation 権限の設定
Amazon Athena のテーブルをクエリするために必要なアクセス権限を Lake Formation 内で付与する手順は次のとおりです。
- AWS Lake Formation コンソールに移動し、IAM ロールが管理者として追加されていることを確認します (図 2 を参照)。
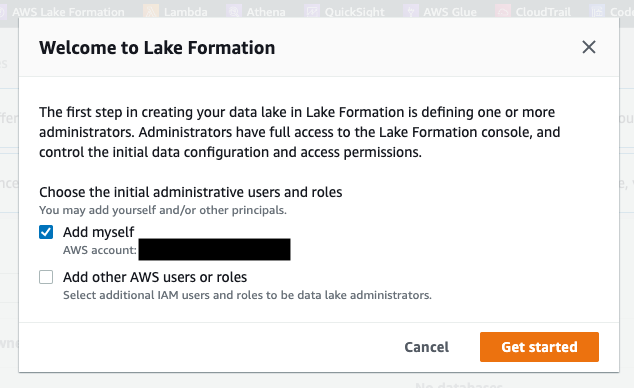 図 2: AWS Lake Formation に管理者として IAM ロールを追加する
図 2: AWS Lake Formation に管理者として IAM ロールを追加する
- [Create Database] (データベースの作成) の画面で、左側の [Database] (データベース) をクリックし、
omicsdbというデータベースを作成します。このデータベースにバリアントテーブルとアノテーションテーブルを追加します (図 3 参照)。
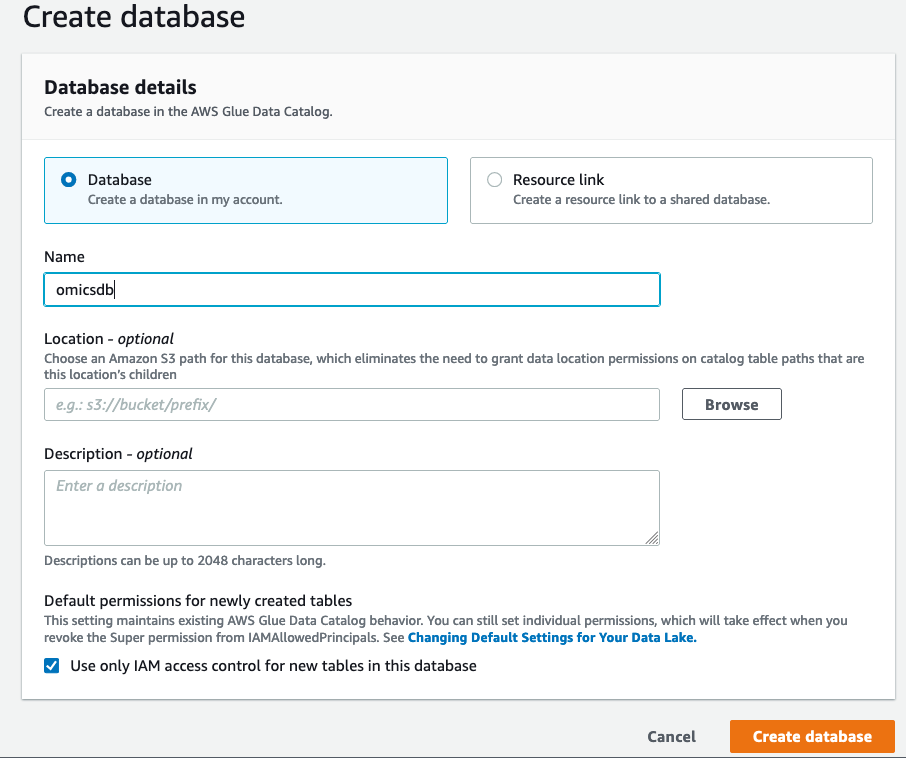 図 3: データベースを作成してバリアントテーブルとアノテーションテーブルを追加する
図 3: データベースを作成してバリアントテーブルとアノテーションテーブルを追加する
- コンソールの左側にある [Table] (テーブル) をクリックします。
omicsvariantstoreを選択し、[Actions] (アクション) で [Create Resource Link] (リソースリンクの作成) を選択します(図 4 を参照)。
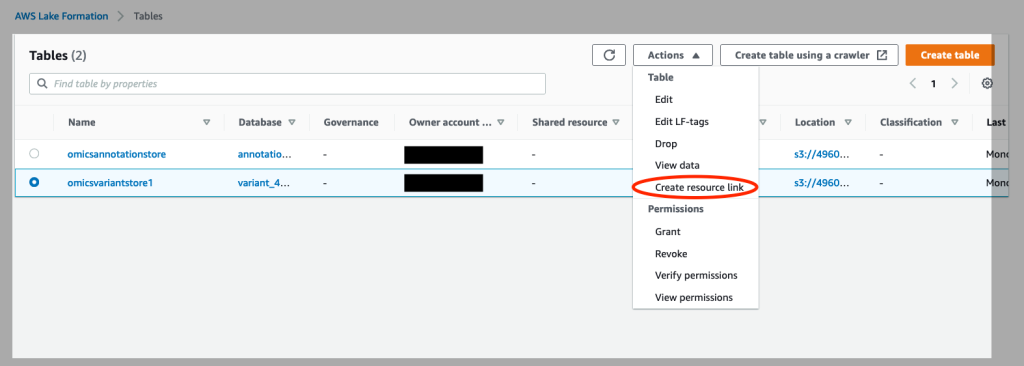 図 4: AWS Lake Formation のオミックスアノテーションストアとオミックスバリアントストアのリソースリンクを作成する
図 4: AWS Lake Formation のオミックスアノテーションストアとオミックスバリアントストアのリソースリンクを作成する
- [Create Resource Link] (リソースリンクの作成) の画面で、リソースリンクに名前 (たとえば
omicsvariants) を付け、図 2 で作成したデータベース (omicsdb) を選択します。
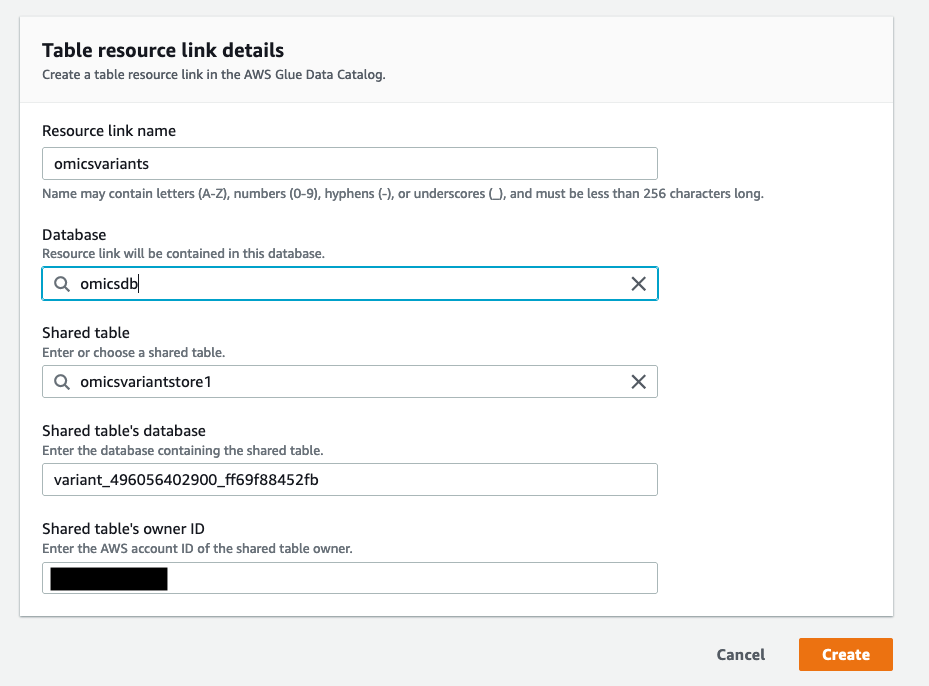 図 5: Omics バリアントストアのリソースリンクを作成し新しく作成したデータベース
図 5: Omics バリアントストアのリソースリンクを作成し新しく作成したデータベースomicsdbに追加する
-
- 手順 3 と 4 を繰り返して
omicsannotationstoreを作成します。
- 手順 3 と 4 を繰り返して
Amazon Athena でバリアントとアノテーションをデータベースとして使用できるようになったので、Athena コンソールで SQL クエリを使用してクエリを実行できます。これらのクエリの Athena ワークグループは Athena バージョン 3 クエリエンジンを使用する必要があることに注意してください。
Amazon SageMaker (SageMaker) ノートブックを使用してバリアントをクエリするには、バリアントテーブルとアノテーションテーブルをクエリするためのアクセス権限を SageMaker ノートブックインスタンスロールに付与するという追加のステップがあります。
- AWS Lake Formation コンソールに移動し、[Tables] (テーブル) をクリックします。
- AWS Lake Formation 権限にあるバリアントのリソースリンクを選択してください。
- アクションで [Grant on Target] (ターゲットに付与) を選択します。
- [Grant Permissions screen] (権限の付与画面)で、ノートブックが使用する IAM プリンシパルとして SageMaker 実行ルールを選択します。
- [Table Permissions] (テーブル権限) で [Select] と [Describe] を選択します。
- その他の選択項目は、図 6 に示すようにデフォルト設定のままにします。
omicsannotationsリソースリンクの Amazon SageMaker ノートブックインスタンスロールについて、ここで説明したステップ 1 ~ 6 を繰り返します。
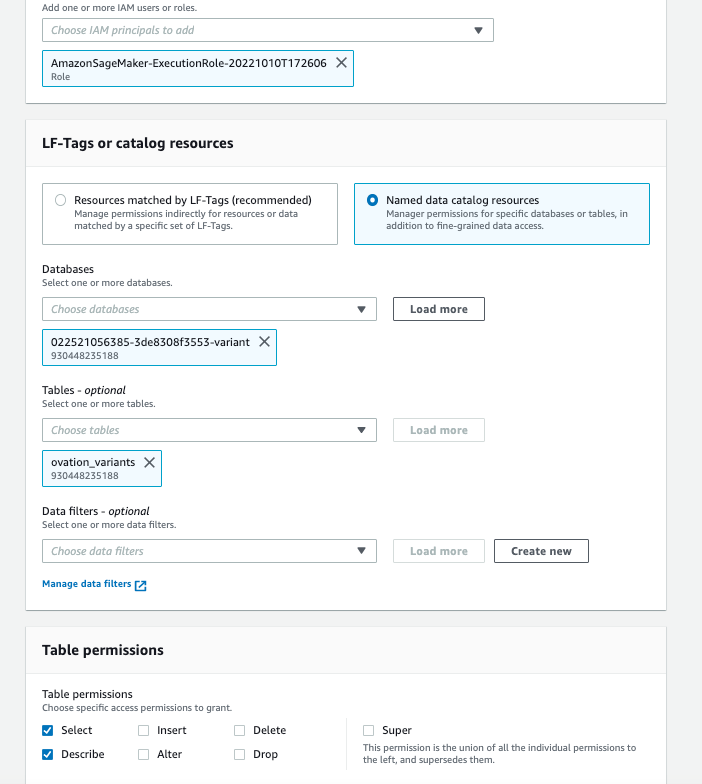 図 6: オミックスバリアントとアノテーションリソースリンクの SageMaker Notebook 実行ロールに対する AWS Lake Formation アクセス権限
図 6: オミックスバリアントとアノテーションリソースリンクの SageMaker Notebook 実行ロールに対する AWS Lake Formation アクセス権限
クエリ例が記載された Jupyter ノートブックはこちらから入手できます。このサンプルノートブックでは、AWS SDK for Pandas を使用して Athena に接続してクエリを実行します。
バリアントからインサイトまで
データレイクにてマルチオミックス及びマルチモーダルデータを活用しているお客様は、これらのデータセットと新たに追加されたゲノムバリアントを結合してクエリし、インサイトを得ることができます。この例では AWS Data Exchange で入手可能な Ovation Dx 非アルコール性脂肪肝疾患 (NAFLD) データセットサンプルから全ゲノムシーケンスバリアントと患者人口統計データを使用しました。ClinVar のバリアントアノテーションを使用して、クエリを実行してこの 10 人の患者のコホートからインサイトを得る方法を示します。
10 人の患者からの 30X カバレッジの全ゲノムシーケンスデータがオミックスシーケンスストアにインポートされ、データをバリアントに変換するために GATK ベストプラクティスに基づく WDL ワークフローが開始されます。生成されたバリアントはバリアントストアにインポートされ、ClinVar VCF はオミックスアノテーションストアにインポートされ、どちらも AWS Lake Formation のテーブルとして表示されます。
患者情報は AWS Glue Data Crawler でクロール可能な CSV ファイル形式であり、テーブルとして提供されます。患者情報とバリアントおよびバリアントアノテーションを結合することで患者のコホートへのインサイトを得ることができます。
 図 7 : ゲノムデータと臨床データを使用したインサイトの視覚的表現の例
図 7 : ゲノムデータと臨床データを使用したインサイトの視覚的表現の例
例えば図 7 は患者毎に病原性のある遺伝子や病気を引き起こす可能性が高い病原性バリアントをもつ遺伝子を示しています。興味深いことに NAFLD と診断されたこの患者コホートでは、すべての患者が家族性高コレステロール血症のリスクを高める APOA2 遺伝子のバリアントを持っています。NAFLD と高コレステロール値および肥満との間に関連性があるという幾つかのエビデンスがあります。このコホートの患者の何人かは 1 型糖尿病に罹患しやすくなるバリアントも持っています。
この例は、ゲノムバリアントを臨床アノテーションと臨床表現型と組み合わせることで新しいインサイトを得る方法と Amazon Omics がゲノムデータに対してこれをどのように合理化するかを示しています
結論
このブログ投稿では次の方法を示しました。
- オミックスシーケンスストアへのシーケンスデータの取り込みのエンドツーエンドプロセスの自動化
- オミックスワークフローを使用した事前定義されたワークフローを通じたデータ分析
- オミックスバリアントストアを使用した、データレイク内の他のモダリティのデータと共に後工程の分析に使用可能なクエリ対応のゲノムバリアントの生成
さらに、Amazon Athena と Amazon SageMaker を使用して、Amazon Omics に保存されているゲノムバリアントとアノテーションデータをユーザのデータレイクの臨床データと結合して分析する方法も示しました。
このアーキテクチャにより、お客様は運用上のオーバーヘッドを最小限に抑え、インサイトを得るまでの時間を短縮する、再現可能で自動化されたエンドツーエンドのインフラストラクチャをデプロイできます。
サンプルコードは GitHub で入手できます。GitHub リポジトリ内の README ファイルにはセットアップ方法に関する詳細な手順が記載されています。このサンプルコードはデモンストレーションを目的としており本番環境で使用するためのものではないことに注意してください。
ご利用のリージョンでのサービスの提供状況や料金の詳細は Amazon Omics 製品ページをご確認ください。
AWS がお客様のために何ができるかについては、AWS の担当者にお問合せください。
翻訳は Solutions Architect 岡田が担当しました。原文はこちらです。