Amazon Web Services ブログ
MIMIC-III データと Amazon Athena を使用して、データベースなしでバイオメディカルインフォマティクスを実行
バイオメディカル研究者は、正確で詳細なデータにアクセスする必要があります。MIT MIMIC-III データセットは人気のあるリソースです。Amazon Athena を使用すると、最初にデータをデータベースにロードすることなく、MIMIC-III に対して標準 SQL クエリを実行できます。分析は常に MIMIC-III データセットの最新バージョンを参照します。
この投稿では、Athena で MIMIC-III データセットを使用可能にし、AWS の MIMIC-III 分析環境への自動アクセスを提供する方法について説明します。また、従来のデータベースを使用した MIMIC-III リファレンスバイオインフォマティクス研究と、Athena を使用した同じ研究を比較します。
概要
長期にわたって多くの患者のさまざまな測定値をキャプチャするデータセットは、研究の発見と改善された臨床的判断に分析と機械学習を用いて推進できます。これらの機能は、MIT 計算生物学研究所 (LCP) MIMIC-III データセットについて記述しています。LCP 研究者の発言:
「MIMIC-III は、2001 年から 2012 年にかけてベスイスラエルメディカルセンターの救命治療室に入院した患者約 6 万人の匿名化された健康関連データを含む、大規模な公開データベースです。MIMIC は、疫学、臨床的判断ルールの改善、電子ツールの開発に及ぶさまざまな分析研究をサポートしています。中でも 3 つの要因に注目したいと思います。まず、公開的かつ自由に利用可能で、ICU 患者の多様で大規模な集団を含みます。そして実験結果、電子文書、ベッドサイドモニターの傾向と波形を含む高時間解像度データを包含します」
最近、AWS の Registry of Open Data (RODA) プログラムにより、AWS クラウドから MIMIC-III データセットを利用できるようになりました。これで、MIMIC-III データセットをダウンロード、コピーせずに、または保存費用を支払わずに使用できます。代わりに、Amazon EC2、Athena、AWS Lambda、または Amazon EMR などの AWS サービスを使用して、AWS クラウドの MIMIC-III データセットを分析できます。AWS クラウドの可用性によって、より迅速で安いデータセット調査が可能になります。
Athena などのサービスは、MIMIC-III データセットに対する新しい分析アプローチも提供します。Athena を使用すると、最初にデータをデータベースにロードすることなく、MIMIC-III に対して標準 SQL クエリを実行できます。RODA プログラムでホストされている MIMIC-III データセットを参照できるため、分析では常に MIMIC-III データセットの最新バージョンを参照します。ライブホスティングで事前にかかる時間と労力を削減し、データの同期問題を解消し、データ分析を改善して、全体的な調査コストを削減します。
MIMIC-III データの変換
歴史的に、MIMIC チームは、MIMIC-III データセットを圧縮した (gzip 圧縮) CSV 形式で配布してきました。CSV 形式を選択したことは、分析のために MIMIC-III データを従来のリレーショナルデータベースにロードすることを反映しています。
対照的に、MIMIC チームは MIMIC-III データセットを次の形式で RODA プログラムに提供します。
- 従来の CSV 形式
- Athena などの最新のデータ処理テクノロジーに向けて最適化された Apache Parquet 形式
Apache Parquet は列ごとにデータを保存するため、特定の列を取得するクエリは、テーブル全体を読み取らずに実行することができます。この最適化により、バイオメディカルインフォマティクスに共通する多くのクエリタイプのパフォーマンスが向上し、コストが削減されます。
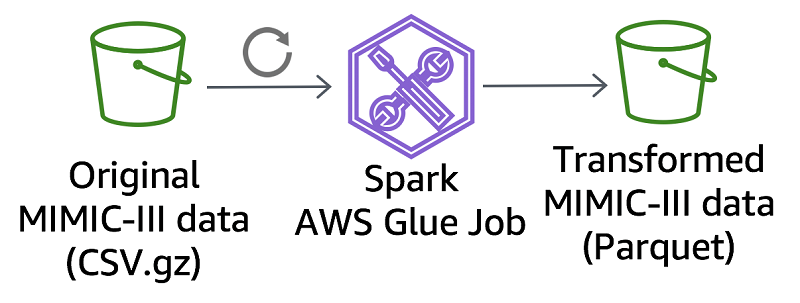
元の MIMIC-III CSV データセットを Apache Parquet に変換するために、AWS Glue を使用してデータ変換ジョブを作成しました。MIMIC チームは必要に応じて AWS Glue ジョブを実行するようにスケジュールし、CSV データセットへの変更によって RODA プログラムの Parquet ファイルを更新します。スケジュールされたこれらの更新は、MIMIC チームの CSV データセット作成手順を妨げることなく、Parquet ファイルを最新状態に保ちます。mimic-code GitHub リポジトリでこの AWS Glue ジョブのコードを見つけます。同じコードをベースとして使用して、他の CSV から Parquet への変換を開発します。
MIMIC-III NOTEEVENTS テーブルを変換するコードは、貴重なリソースを提供します。このテーブルには、コンマ、二重引用符、および変換が困難になる可能性のある他の文字を含め、複数行で構成される長い医療メモが保存されます。たとえば、次の Spark 読み取りステートメントを使用して、その CSV ファイルを解釈できます。
MIMIC-III を使用した留置動脈カテーテル研究の実行
たとえば、MIMIC チームは、MIMIC 留置動脈カテーテル (IAC) の動脈圧ライン研究コードを提供しています。この研究では、MIMIC-III データセットを使用して、公開された研究血行動態的に安定した呼吸不全患者における留置動脈カテーテルと死亡率との関連性から発見した結果を再現しています。
GitHub の MIT 計算生物学研究所 MIMIC コードリポジトリで、動脈圧ライン研究コードと、その他多くの分析例を入手できます。提供されたコードの詳細については、JAMA 論文 MIMIC コードリポジトリ: 救命治療研究における再現性の実現を参照してください。
動脈圧ライン研究コードは、約 1,400 行の SQL で構成されています。PostgreSQL データベース用に開発して、実行し、さらに Python および R コードで分析します。PostgreSQL の代わりに Athena を使用して Parquet MIMIC-III データセットに対して動脈圧ライン研究を実行し、次の質問に答えました。
- PostgreSQL の代わりに Athena を使用して研究を実行するには、どのような修正が必要ですか?
- パフォーマンスはどう違いますか?
- 費用はどう違いますか?
研究で使用された一部の SQL 機能は、Athena での動作と PostgreSQL での動作で異なります。その結果、SQL ステートメントの約 5% を変更しなければなりませんでした。具体的に SQL ステートメントを Athena で使用するためには、次のタイプの変更が必要です。
マテリアライズドビューを使用する代わりに、Athena で新しいテーブルを作成します。たとえば、CREATE MATERIALIZED VIEW ステートメントは CREATE TABLE として書き込むことができます。
- Athena で
SET SEARCH_PATHなどのスキーマコンテキストステートメントを使用する代わりに、参照されるテーブルのスキーマを明示的に宣言します。たとえば、クエリの前にSET SEARCH_PATHステートメントを設定し、次のようにスキーマなしでテーブルを参照する代わりに、
SET SEARCH_PATH TO mimiciii;
left join chartevents ce
すべてのテーブルリファレンスの一部としてスキーマを宣言します。
left join mimiciii.chartevents ce
- エポックタイムとタイムスタンプ演算は、Athena では PostgreSQL と異なる動作を行います。そのため、算術演算の代わりに、date_diff() を使用して、戻り値として秒単位の時間を指定します。
extract(epoch from endtime-starttime)/24.0/60.0/60.0
上記のステートメントは次のように記述できます。
date_diff('second',starttime, endtime)/24.0/60.0/60.0
- Athena は、「数値」データ型の代わりに「ダブル」データ型を使用します。
ROUND( cast(f.height_first as numeric), 2) AS height_first,
上記のステートメントは次のように記述できます。
ROUND( cast(f.height_first as double), 2) AS height_first,
- VARCHAR フィールドが空の場合、NULL として検出されません。代わりに、VARCHAR を空の文字列と比較します。たとえば、次のようなフィールドを検討します。
where ce.value IS NOT NULL
ce.value が VARCHAR の場合、次のように書き込むことができます。
where ce.value <> ''
動脈圧ライン研究 SQL ステートメントにこれらの小さな編集を行った後、研究は PostgreSQL の代わりに Athena を使用して実行されます。両方法とも生成される出力は同じです。
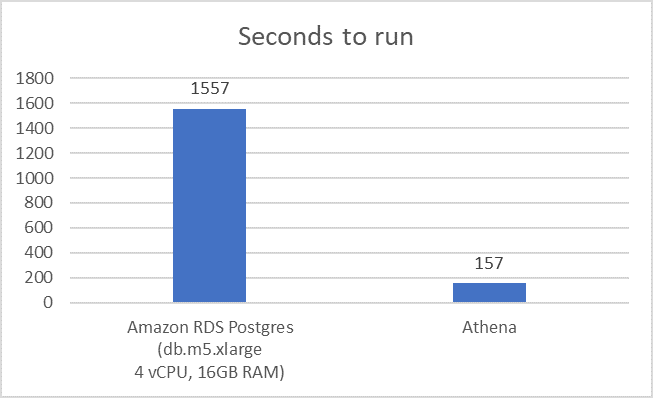
次に、動脈圧ライン研究の分析において、Athena と PostgreSQL の速度を比較します。次のグラフに示すように、Parquet 形式 MIMIC-III データセットの Athena クエリを使用した動脈圧ライン研究は、Amazon RDS PostgreSQL データベースでの同じデータのクエリより 10 倍高速で実行されます。
Athena で動脈圧ライン研究を実行するコストと、PostgreSQL で実行するコストを比較します。RDS PostgreSQL データベースは、データベースの実行時間に対して 1 秒単位で請求されますが、Athena クエリは、各クエリでスキャンするデータのギガバイトごとに請求されます。根本的な料金の違いのため、コストを比較するにはいくつかの仮定が必要です。
RDS PostgreSQL で動脈圧ライン研究を実行するには、次の手順が必要であり、実働 8 時間がかかると想定できます。
- 新しい RDS PostgreSQL データベースをデプロイします。
- MIMIC-III データセットをロードします。
- Jupyter ノートブックを接続します。
- 動脈圧ライン研究を実行します。
- RDS データベースを終了します。
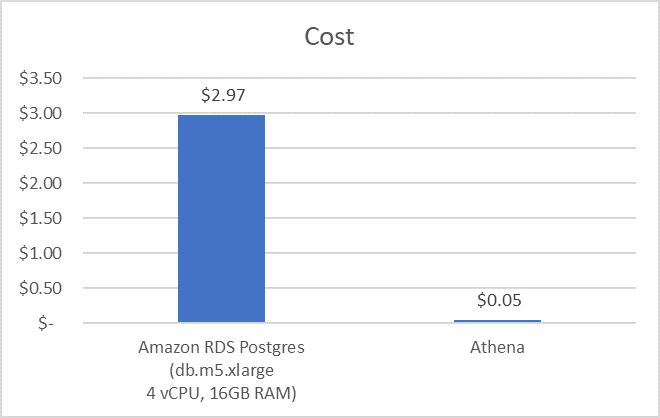
次に、RDS PostgreSQL データベースを 8 時間実行するコスト (1 時間あたり 0.37 USD、100 GB EBS ストレージで db.m5.xlarge) と、Athena を使用するデータセットに対して動脈圧ライン研究を実行するコスト (0.005 USD/GB でスキャンしたデータのうち 9.93 GB) を比較します。次のグラフが示すように、ほぼ 60 倍のコスト削減を実現できます。
AWS での MIMIC-III の操作
MIMIC-III は、患者の臨床ケアに関する詳細情報を含め、公的に利用可能なデータセットです。このため、MIMIC チームでは、アクセスする前にトレーニングコースを完了し、正式なリクエストを送信する必要があります。詳しくは、アクセスのリクエストを参照してください。
MIMIC-III データセットへのアクセスを取得したら、PhysioNet ウェブサイトにログインし、プロファイル設定で AWS アカウント ID を指定します。次に、MIMIC-III 臨床データベースページの [ファイル] にある [アクセスリンクのリクエスト] をクリックします。 その後、AWS の MIMIC-III データセットにアクセスできます。

以下の [スタックを起動] を選択して、AWS アカウントで MIMIC-III データセットの操作を開始します。これにより、AWS CloudFormation テンプレートが起動され、MIMIC-III Parquet 形式でのデータセットのインデックスが AWS Glue データカタログにデプロイされます。また、動脈圧ライン研究コードと、MIMIC が提供したその他多くの分析例を事前にロードした Amazon SageMaker ノートブックインスタンスをデプロイします。
AWS CloudFormation テンプレートがデプロイされたら、[出力] を選択し、リンクに従って Jupyter ノートブックにアクセスします。そこから、ファイルシステムを参照してパス ./mimic-code/notebooks/aline-aws/aline-awsathena.ipynb にアクセスし、動脈圧ライン研究コードを開いて実行できます。
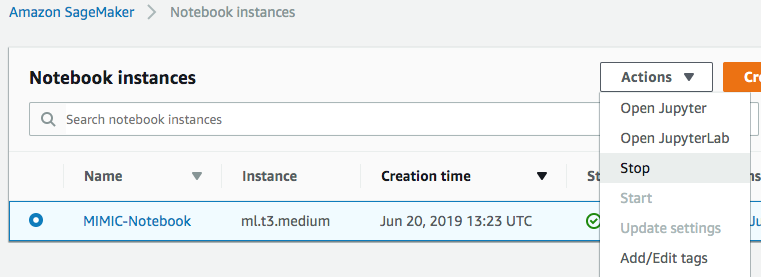
分析が完了したら、Jupyter ノートブックインスタンスを停止して、コンピューティング料金を一時停止できます。 作業を続けたいときはいつでも、インスタンスを開始して、中断したところから続行します。
まとめ
RODA プログラムは、MIMIC-III データセットのグローバルなバイオメディカル研究リソースへの迅速で安いアクセスを提供します。AWS Glue や Athena のようなクラウド固有の分析ツールは研究を加速させ、MIT 計算生物学研究所のようなグループは Apache Parquet のような最新データ形式でデータの可用性を開拓しています。
この投稿で示された分析アプローチとコードは、一般に適用され、俊敏性を高めてコストを削減します。既存のバイオメディカルインフォマティクス研究を強化するか、新しいバイオメディカルインフォマティクス研究を実行する際に、それらの使用を検討してください。
著者について
 James Wiggins は AWS のシニアヘルスケアソリューションアーキテクトです。 彼は組織が世界の医療にポジティブな影響を与えるようにテクノロジーを使用することに情熱をもっています。彼はまた、妻と 3 人の子供と余暇を楽しんでいます。
James Wiggins は AWS のシニアヘルスケアソリューションアーキテクトです。 彼は組織が世界の医療にポジティブな影響を与えるようにテクノロジーを使用することに情熱をもっています。彼はまた、妻と 3 人の子供と余暇を楽しんでいます。
 Alistair Johnson はリサーチサイエンティストで、マサチューセッツ工科大学出身です。Alistair は、MIMIC-III データセットと eICU-CRD データセットを公開しており、ICU データの操作に対して豊富な経験と専門知識を備えています。現在、彼は臨床疫学と意思決定を支援するための機械学習に焦点を当てて研究を行っています。
Alistair Johnson はリサーチサイエンティストで、マサチューセッツ工科大学出身です。Alistair は、MIMIC-III データセットと eICU-CRD データセットを公開しており、ICU データの操作に対して豊富な経験と専門知識を備えています。現在、彼は臨床疫学と意思決定を支援するための機械学習に焦点を当てて研究を行っています。
