計算化学の分野の Machine Learning (ML) メソッドは、急速に成長しています。アクセスが容易なオープンソルバー (TensorFlow と Apache MXNet など)、ツールキット (RDKit 化学情報ソフトウェアなど)、オープン科学イニシアチブ (DeepChem など) は、毎日の研究でこれらのフレームワークを容易に使用できるようにします。科学情報分野では、多くのアンサンブル計算化学ワークフローが多数の化合物を消費し、さまざまな記述子特性をプロファイリングする能力を必要とします。
このブログ投稿では、2 段階ワークフローについて説明します。最初のステージでは、約 1100 の候補の分子を採取し、AWS Batch を使用して、Dockerized RDKit を使用した 2D 分子記述子を計算します。 MoleculeNet.ai – ESOLV からの元のデータセットには、各化合物の測定済みの logSolubility (mol/L) が含まれます。第 2 ステージでは、Amazon SageMaker を Apache MXNet で使用して、線形回帰予測モデルを作成します。ML モデルはトレーニングと検証の 70/30 分割を実行し、30 エポック後の RMSE = 0.925 で、適合度 (Rˆ2) は 0.9 になります。
このブログ投稿では、単純化された分子入力ライン入力システム (SMILES) の入力を処理するワークフローを作成し、その後、Amazon SageMaker に送出して、logSolubility を予測するモデルを作成します。
概要
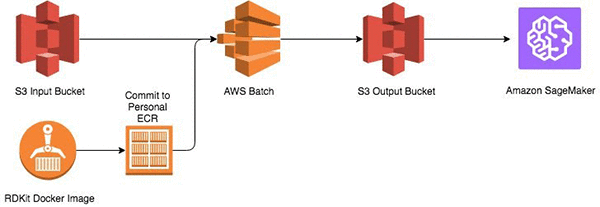
Amazon S3 バケットで、SMILES 構造を保管することから開始します。次に、Amazon Elastic Container Repository (Amazon ECR) イメージを Python ベースの実行スクリプトで構築し、ライブラリをイメージにサポートして、AWS Batch で実行します。計算の出力は、別の S3 バケットに保管され、それが Amazon SageMaker に入力されます。
前提条件
このブログ投稿の手順に従うには、AWS アカウントが必要です。ローカルコンピュータで Docker イメージを作成し、Docker をインストールして、設定します。AWS コマンドラインインターフェース (AWS CLI) をインストールすることも必要です。
ステージ 1: AWS Batch の使用
AWS Batch は Amazon ECR に保管されたコンテナ化されたアプリケーションから実行されているジョブのマネージド型サービスです。ワークフローを実行するために必要なツールをインストールするための基本オペレーティングシステム (OS) を含む「amazonlinux:latest」イメージは、パブリック Docker 登録から取得されました。Docker をインストールした後で、コマンドラインシェルを開き、次のコマンドを実行してください。
docker pull amazonlinux:latest
イメージレイヤーを取得した後で、対話形式のセッションが開始して、AWS Batch での記述子計算のためにイメージを準備します。AWS CLI、RDKit、Boto3 フレームワークパッケージをイメージにインストールします。実行中の Docker コンテナでは、次のコマンドを実行します。
docker run -it amazonlinux:latest
yum install epel-release aws-cli python-pip
pip install boto3
RDKit をインストールするには、まず repo を有効化します。コードについては、EPEL 6 リポジトリを参照してください。
repo を有効にした後で、次のコマンドを実行することによりそれをインストールします。
yum install python2-rdkit
mkdir -p /data
次の Python コードをイメージに追加し、それを /data/mp_calculate_descriptors.py として保存します。
from __future__ import print_function
import os
import sys
import boto3
import argparse
import uuid
import datetime
import subprocess
from rdkit import Chem
import multiprocessing as mp
from multiprocessing import Pool,Process
from rdkit.Chem import Descriptors as desc
# establish an Amazon S3 connection; an IAM role is needed to access Amazon S3
s3=boto3.client('s3')
# change to execution dir
os.chdir("/data")
# execute the main RDKit descriptor worker
def smiles_desc(smiles_str):
output_desc=[]
m = Chem.MolFromSmiles(smiles_str)
output_desc=smiles_str, \
desc.MolWt(m), \
desc.Ipc(m), \
desc.TPSA(m), \
desc.LabuteASA(m), \
desc.NumHDonors(m), \
desc.NumHAcceptors(m), \
desc.MolLogP(m), \
desc.HeavyAtomCount(m), \
desc.NumRotatableBonds(m), \
desc.RingCount(m), \
desc.NumValenceElectrons(m)
desc_shard=str(output_desc).strip('()')
smiles_out(desc_shard,csv_header)
return
# write out the results to the local filesystem
def smiles_out(single_des_out,csv_header):
fd=open("%s_smiles_result.csv" %csv_header,"a")
fd.write(single_des_out)
fd.write('\n')
fd.close
# upload to an s3 bucket
def s3_upload(csv_header):
env_S3_OUT=os.environ['OUTPUT_SMILES_S3']
s3.upload_file("%s_smiles_result.csv" %csv_header,"%s" %env_S3_OUT,"%s_smiles_result.csv" %csv_header)
# calculate stat function
def calc_perf(input_file,timedelta):
calc_perf_stat=int(len(input_file)/timedelta.total_seconds())
return calc_perf_stat
if __name__ == "__main__":
# accepted args for command line execution
parser = argparse.ArgumentParser(description='Process SMILES')
parser.add_argument("-i", dest="smiles", required=False, help='SMILES string')
parser.add_argument("-f", dest="smiles_file", required=False, help="File for Batch SMILES processing")
args = parser.parse_args()
smiles_str=args.smiles
smiles_files=args.smiles_file
csv_header=str(uuid.uuid4())
# if defined on the command line
if smiles_str:
print (smiles_desc(smiles_str))
smiles_out(str(smiles_desc(smiles_str)),csv_header)
s3_upload(csv_header)
# if importing a file
# by default, parallelism expands to available cores exposed to the container
elif smiles_files:
infile=open(smiles_files,"r")
pool = mp.Pool()
start_calc=datetime.datetime.now()
smiles_list=list(infile)
pool.map(smiles_desc,smiles_list)
pool.close()
end_calc=datetime.datetime.now()
delta_calc=end_calc-start_calc
stat_calc=calc_perf(smiles_list,delta_calc)
print ("number of structures/sec: %s"%stat_calc)
s3_upload(csv_header)
# if file is defined through env
# by default, parallelism expands to available cores exposed to the container
elif os.getenv('INPUT_SMILES'):
env_SMILES=os.environ['INPUT_SMILES']
infile=open(env_SMILES,"r")
pool = mp.Pool()
start_calc=datetime.datetime.now()
smiles_list=list(infile)
pool.map(smiles_desc,smiles_list)
pool.close()
end_calc=datetime.datetime.now()
delta_calc=end_calc-start_calc
stat_calc=calc_perf(smiles_list,delta_calc)
print ("number of structures/sec: %s"%stat_calc)
s3_upload(csv_header)
# if file is in the S3 import bucket, specify an AWS BATCH job def
# by default, parallelism expands to available cores exposed to the container
elif os.getenv('INPUT_SMILES_S3'):
env_S3_SMILES=os.environ['INPUT_SMILES_S3']
s3_dn=subprocess.Popen("aws s3 cp %s /data" %env_S3_SMILES, shell=True)
s3_dn.communicate()
s3_file=env_S3_SMILES.split('/')[-1]
infile=open("/data/%s" %s3_file,"r")
pool = mp.Pool()
start_calc=datetime.datetime.now()
smiles_list=list(infile)
pool.map(smiles_desc,smiles_list)
pool.close()
end_calc=datetime.datetime.now()
delta_calc=end_calc-start_calc
stat_calc=calc_perf(smiles_list,delta_calc)
print ("number of structures/sec: %s"%stat_calc)
s3_upload(csv_header)
else:
print ("No SMILES FOUND...")
sys.exit(0)
このコードは、記述子を計算するためのメインエンジンです。このスクリプトは、S3 バケットから入力 SMILES ファイルを読み取り、一連の記述子を計算し、別の S3 バケットに結果を保管します。
Amazon S3 コンソールを開き、 rdkit-input-<initials> と rdkit-processed-<initials> と呼ばれるバケットを AWS アカウントにアクセスを制限して作成します。
次に、AWS アカウントで Amazon ECR 登録に Docker イメージをコミットします。Amazon ECS コンソールを開き、左パネルの [Repositories (リポジトリ)] を選択し、[Create repository (リポジトリの作成)] を選択することで新しい登録を作成します。次のようなエンドポイントが得られます。
<account_number>.dkr.ecr.us-east-1.amazonaws.com/awsbatch/rdkit
エンドポイントを AWS Batch にアクセス可能にするには、Docker イメージをエンドポイントにプッシュします。
# Get the temporary login credentials of your registry.
aws ecr get-login —no-include-email
# Execute the login command from "aws ecr get-login".
docker login <params>
# Commit the local container to a new docker image.
docker commit -m "setting up rdkit infra" <containerid> amazonlinux:rdkit
# Tag the new image with your Amazon ECR tag created in the Amazon ECS console.
docker tag amazonlinux:rdkit <account_number>.dkr.ecr.us-east-1.amazonaws.com/awsbatch/rdkit:latest
# Push the image to your private registery.
docker push <account_number>.dkr.ecr.us-east-1.amazonaws.com/awsbatch/rdkit:latest
AWS Batch は、ジョブ定義を使用して設定することが必要で、その後、コンピューティング環境で実行されるジョブキューに送信されます。JSON ジョブ定義は、RDKit ジョブの入力パラメーターを指定します。以下は、このワークフローの例です。
{
"jobDefinitionName": "rdkit1",
"jobDefinitionArn": "arn:aws:batch:us-east-1:<account_number>:job-definition/rdkit1:1",
"revision": 1,
"status": "ACTIVE",
"type": "container",
"parameters": {},
"retryStrategy": {
"attempts": 1
},
"containerProperties": {
"image": "<account_number>.dkr.ecr.us-east-1.amazonaws.com/awsbatch/rdkit:latest",
"vcpus": 4,
"memory": 2000,
"command": [
"python",
"/data/mp_calculate_descriptors.py"
],
"jobRoleArn": "arn:aws:iam::<account_number>:role/BatchS3Role",
"volumes": [],
"environment": [
{
"name": "OUTPUT_SMILES_S3",
"value": "rdkit-processed"
},
{
"name": "INPUT_SMILES_S3",
"value": "s3://rdkit-input/deepchem.smiles"
}
],
"mountPoints": [],
"ulimits": []
}
}
ジョブ定義で、OUTPUT_SMILES_S3 と INPUT_SMILES_S3 環境変数を定義します。これは、Amazon S3 にアップロードされる SMILES ファイルへのパスです。この変数は、コンテナの Python スクリプトに渡されます。正しいアクセス権限があることを確認するために、Amazon S3 への読み書きアクセスをもつ jobRole (IAM コンソールで設定) を定義してください。 Python スクリプトは、本来、並列化されているため、インスタンスが大きいほど、SMILES ファイルを処理するのにより大きなレベルの並列化が行われます。下表は EC2 インスタンスの c4 と m4 ファミリを使用するデータセットをプロファイリングします。

AWS Batch ジョブを実行します。Rdkit-processed バケットで、以下に類似するファイル (*_smiles_result.csv) が表示されます。
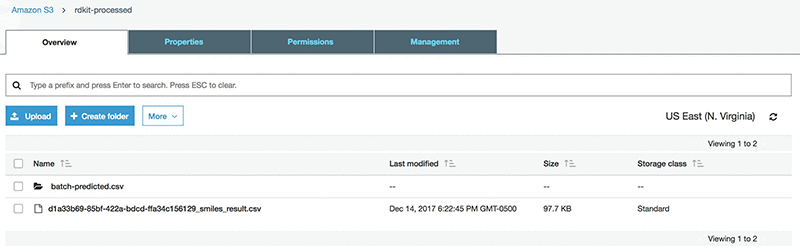
元の入力ファイルには、測定済みの logSolubility (mol/L) の列が含まれます。SMILES をプライマリキーとして使用する Amazon SageMaker ステージを準備するために、この列を結果ファイルにアペンドします。これは、S3 から csv をダウンロードし、測定済みの logSolubility 値をアペンドした後で、再びそれをアップロードすることによって行うことができます。
第 2 ステージ: Amazon SageMaker の使用
Amazon SageMaker コンソールで、[Dashboard (ダッシュボード)] の下で、[Create notebook Instance (ノートブックインスタンスの作成)] を選択し、以下のスクリーンショットに示されたとおり、インターフェイスの詳細を入力します。コンソールを初めて使用する場合、Amazon SageMaker は Amazon S3 にアクセスするために必要な IAM ロールを作成するように求めます。VPC とサブネット設定を設定することはオプションです。
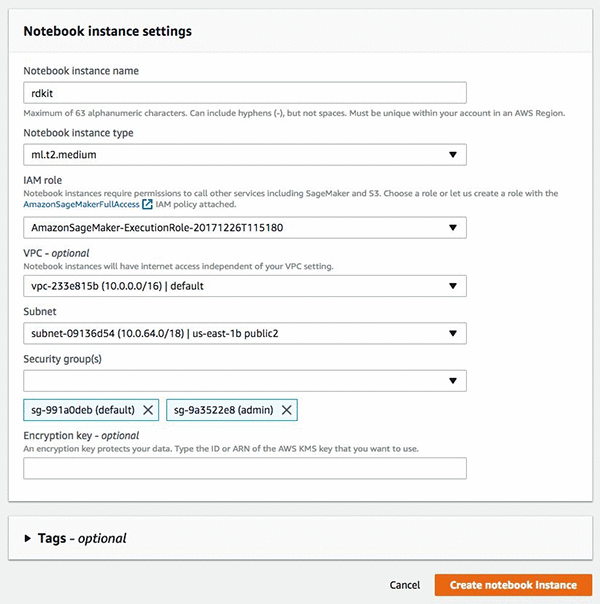
これらのタスクを完了した後で、[Create notebook instance (ノートブックインスタンスの作成)] を選択すると、数分後にインスタンスが作成されます。インスタンスが開始した後で、[Open (開く)] を選択すると、インスタンスと Jupyter ノートブックインターフェイスにリダイレクトされます。
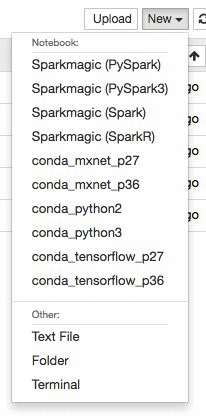
conda_mxnet_p27 環境を使用して、インスタンスで新しい Jupyter ノートブックを作成します。また、TensorFlow のみならず、Python 2 と Python 3 環境を使用する環境も提供します。代わりに、単にここでノートブック全体をダウンロードすることもできます。
候補の化合物のトレーニングのためのノートブックを作成しましょう。まず、溶解度の値を追加した後で、ワークフローの最初の部分から結果ファイルを保管する場所にいくつかの S3 バケット変数を定義する必要があります。
#SET ENVIROMENT VARIABLE
#Set the bucket name for the processed bucket from AWS-Batch
#Set the key name for the output csv file from AWS-Batch
BUCKET_NAME="rdkit-processed"
KEY_NAME="final_sol.csv"
次に、RDKit ライブラリを環境にインポートする必要があります。
#Install RDKit through conda
!conda install -y -c rdkit rdkit
いくつかの依存関係がインストールされます。次に、この演習を使用するモジュールをインポートします。
import os
import sys
import csv
import urllib
import boto
import subprocess
import numpy as np
import mxnet as mx
from rdkit import Chem
from rdkit.Chem import Draw
import multiprocessing as mp
import matplotlib.pyplot as plt
from ipywidgets import widgets
from IPython.html.widgets import *
from matplotlib.gridspec import GridSpec
from rdkit.Chem.Draw import IPythonConsole
from multiprocessing import Pool,Process
from rdkit.Chem import Descriptors as desc
次に、deepchem GitHub からファイルを読み込みます。
# Download File
urllib.urlretrieve ("https://raw.githubusercontent.com/deepchem/deepchem/master/datasets/delaney-processed.csv", \
"delaney-processed.csv")
では、ファイルから SMILES を読み込み、構造体を解析します。
# Import File
infile=open("delaney-processed.csv","rb")
reader = csv.DictReader(infile)
smiles_list=list(reader)
m=[]
for i in range(len(smiles_list)):
m.append(Chem.MolFromSmiles(smiles_list[i]['smiles']))
print "Number of Structures in file: %s" %len(m)
deepchem set (オプション) で構造体をいくつか視覚化できます。
#Draw a few structures
Draw.MolsToGridImage(m[:8],molsPerRow=4,subImgSize=(200,200),legends=[smiles_list[i]['Compound ID'] \
for i in xrange(len(m[:8]))])
次のような出力が得られます。
次に、前述の AWS Batch ワークフローから結果をインポートします。
# Import our result csv file from the AWS Batch workflow
conn=boto.connect_s3()
bucket=conn.get_bucket(BUCKET_NAME)
key=bucket.get_key(KEY_NAME)
key.get_contents_to_filename(KEY_NAME)
batch_file=open(KEY_NAME,"rb")
batch_reader = csv.reader(batch_file)
out_desc_all=list(batch_reader)
out_desc = [x[1:13] for x in out_desc_all]
データをシャッフルして 70/30 トレーニングと検証セットに分割し、モデリングの準備をします。出力はトレーニングと検証セットのデータシェイプを印刷します。
# Define our input data arrays
out_desc_array=np.array(out_desc,dtype=np.float32)
np.random.shuffle(out_desc_array)
all_data = out_desc_array[:,0:11]
all_label = out_desc_array[:,11:12]
x_train,x_label, y_test,y_label = out_desc_array[:790,0:11], \
out_desc_array[:790,11:12], \
out_desc_array[790:,0:11], \
out_desc_array[790:,11:12]
print np.shape(all_data)
print np.shape(all_label)
print np.shape(x_train)
print np.shape(x_label)
print np.shape(y_test)
print np.shape(y_label)
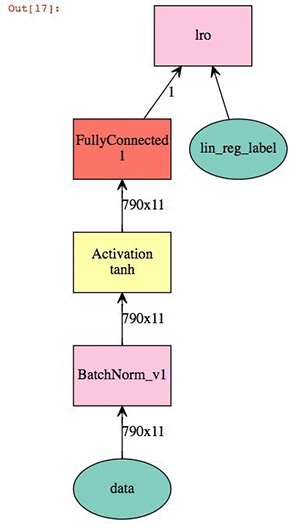
では、線形モデリングパラメーターを定義します。これは、双曲線接線活性化を伴う 2D 記述子セットの比較的単純な線形回帰結合一括正規化です。この出力は、神経系の視覚的な表示です。
# Define Model Parameters
import logging
logging.getLogger().setLevel(logging.DEBUG)
batch_size=20
train_iter = mx.io.NDArrayIter(x_train,x_label, batch_size, shuffle=True,label_name='lin_reg_label')
eval_iter = mx.io.NDArrayIter(y_test, y_label, batch_size, shuffle=False)
all_iter = mx.io.NDArrayIter(all_data, all_label, batch_size, shuffle=False)
train_iter.__dict__
X = mx.sym.Variable('data')
Y = mx.symbol.Variable('lin_reg_label')
fc1 = mx.symbol.BatchNorm_v1(data=X, name="BatchNorm_v1")
tanh1 = mx.symbol.Activation(fc1, act_type="tanh", name="tanh1")
fc2 = mx.sym.FullyConnected(data=tanh1, name='fc2', num_hidden = 1)
lro = mx.sym.LinearRegressionOutput(data=fc2, label=Y, name="lro")
shape={"data": (batch_size,790, 11)}
mx.viz.plot_network(lro,shape = shape)
この時点で、モデルをトレーニングして、テストに対して検証スコアを確認できます。
# Train our model
model = mx.mod.Module(
symbol = lro ,
context = mx.cpu(),
data_names=['data'],
label_names = ['lin_reg_label']# network structure
)
model.bind(data_shapes=[train_iter.provide_data[0]], label_shapes=[train_iter.provide_label[0]])
model.init_params()
model.fit(train_iter, eval_iter,
optimizer='sgd',
optimizer_params={'learning_rate':0.01, 'momentum':0.9},
num_epoch=50,
eval_metric='rmse',
batch_end_callback = mx.callback.Speedometer(batch_size, 10))
およそ 1 秒間に 2 万個のサンプルの速度でトレーニングのデバッグログを取得する必要があります。最後に、結果スコアをプロットし、モデルを使用してデータセット全体を予測することができます。
# Plot our metrics - full cross validation
%matplotlib inline
from scipy import stats
all_preds=model.predict(all_iter).asnumpy()
eval_preds=model.predict(eval_iter).asnumpy()
plt.figure(figsize=(6,6), dpi=80)
plt.scatter(all_label,all_preds, label='All', c='blue',marker='.', alpha=0.2)
plt.scatter(y_label, eval_preds, label='Test/Evaluation', c='green',marker='.')
plt.xlim(-12,3)
plt.ylim(-10,2)
plt.title('Predicted vs Measured ESOLV DeepChem Set')
plt.xlabel('Measured logSolubility (mol/L)')
plt.ylabel('Predicted logSolubility (mol/L)')
plt.legend(loc=4)
print "pearson coefficient of all data: %s" %round(float(stats.pearsonr(all_label,all_preds)[0]),3)
print "pearson coefficient of evaluation data: %s" %round(float(stats.pearsonr(y_label,eval_preds)[0]),3)
delta_preds=all_preds-all_label
def plthist(x):
fig = plt.figure(figsize=(6,6), dpi=80)
plt.hist(delta_preds,bins=x,align='mid',range=(-4,4),rwidth=0.8)
x=plt.xlabel('Element-wise Evaluation Prediction-Target')
interact(plthist,x=(2,50,1))
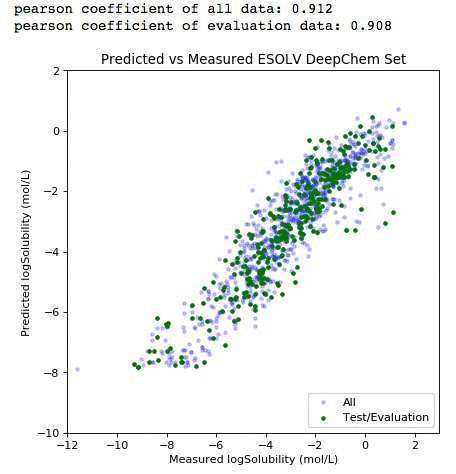
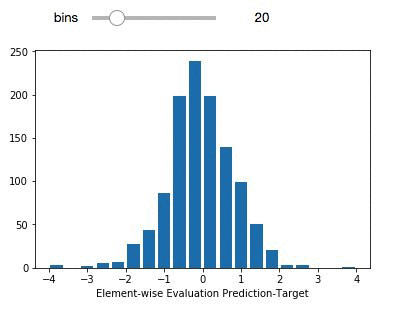
薄紫色のデータポイント (左パネル) は、データセット全体を構成します。緑のサブセットは、独立した検証のために選択された化合物です。評価セットは、90% を超える検証スコアで deepchem セット全体の多様性を適切に表しています。右パネルは、要素ごとの予測誤差分布を表しています。この時点で、構築されたモデルで、エンドポイントを作成して展開することができます。モデルのエンドポイントを展開し、作成する方法の例については、ここを参照してください。
まとめ
このブログ投稿では、Amazon ECS と AWS Batch でコンテナベースの RDKit プラットフォームを作成し、分子記述子計算のための化合物の集合を処理し、Amazon SageMaker を通じて、溶解度を予測するために Apache MXNet ML モデルを開発しました。
さまざまな model.fit() パラメーターを Amazon SageMaker ノートブックで使ってみることをお勧めします。オプティマイザー、学習速度、エポックを変更できます。検証スコアを改善できる場合、ブログのコメント欄にご回答ください。
ご質問があればコメント内に記入してください。
次のステップ
Amazon ML Solutions Lab で Amazon 全体の機械学習の専門家とコネクトしましょう
今回のブログ投稿者について
 Amr Ragab は、AWS のハイパフォーマンスコンピューティングプロフェッショナルサービスコンサルタントで、大規模な計算ワークロードを実行する顧客の支援を専門にしています。余暇には旅行が好きで、技術を日常生活に組み込む方法を見つけています。
Amr Ragab は、AWS のハイパフォーマンスコンピューティングプロフェッショナルサービスコンサルタントで、大規模な計算ワークロードを実行する顧客の支援を専門にしています。余暇には旅行が好きで、技術を日常生活に組み込む方法を見つけています。









 Amr Ragab は、AWS のハイパフォーマンスコンピューティングプロフェッショナルサービスコンサルタントで、大規模な計算ワークロードを実行する顧客の支援を専門にしています。余暇には旅行が好きで、技術を日常生活に組み込む方法を見つけています。
Amr Ragab は、AWS のハイパフォーマンスコンピューティングプロフェッショナルサービスコンサルタントで、大規模な計算ワークロードを実行する顧客の支援を専門にしています。余暇には旅行が好きで、技術を日常生活に組み込む方法を見つけています。