Amazon Web Services ブログ
AWS Batch 上で OpenFold を実行しタンパク質フォールディングのコストを最適化する
この記事は “Optimize Protein Folding Costs with OpenFold on AWS Batch” を翻訳したものです。
この投稿は、OpenFold のリード開発者である Sachin Kadyan 氏と、AWS の AI/ML 担当 Sr. Solutions Architect である Brian Loyal 氏によって寄稿されました。
はじめに
タンパク質の物理的な構造を知ることは創薬プロセスの重要なステップの一つです。AlphaFold v2.0 のような機械学習アルゴリズムは、有用なタンパク質構造を生成するために必要なコストと時間を大幅に削減することができます。またこれらのプロジェクトは、de novo タンパク質設計やタンパク質-リガンド間相互作用解析のための AI 駆動型創薬を加速します。
研究者は、 AlphaFold を使用して 2 億以上のタンパク質構造を公開しています。しかし、より新しいアルゴリズムによってコスト効率や精度が向上する可能性があります。その一例が、AlphaFold に代わる完全なオープンソースの OpenFold であり、広く利用可能な GPU 上で動作するように最適化されています。
この投稿では、以前公開したブログをもとに、 AWS Batch でタンパク質フォールディングのジョブをオーケストレーションする方法を解説します。次に、OpenFold と AlphaFold のパフォーマンスをパブリックな標的タンパク質のデータを用いて比較します。最後に、タンパク質フォールディングのコストを最適化する方法について説明します。
OpenFold とは
科学者はタンパク質の構造を解析するために、アクセスしやすく柔軟性が備えた費用対効果の高いツールを必要としています。OpenFold は、R&D チームにとって幾つかの利点を備えたタンパク質フォールディングモデルです。まず、OpenFold はディープラーニングの研究者に人気のある強力な ML フレームワークである PyTorch を使用しています。次に、コロンビア大学のOpenFold 開発チームは、推論コードや学習コード、モデルの重みをパーミッシブ型オープンソースライセンスで公開しました。さらに開発チームは、OpenFold の学習に使用した 450 万個のタンパク質配列と多重配列アライメント (Multiple sequence alignment; MSA) を、Repository of Open Data on AWS (RODA) を介して共有しました。これは、誰もが制限なくモデルを利用し、改良できることを意味します。最後に、OpenFold は Amazon Elastic Compute Cloud (EC2) の高速コンピューティングインスタンスタイプで使われているような一般的な GPU で効率的に動作するように最適化されています。
OpenFold は、推論中のメモリ使用量を最適化するために、ローメモリでインプレースである Attention 機構などの手法を採用しています。これによりこのアルゴリズムは、40GiB VRAM を搭載した A100 GPU で、最大 4,600 残基の構造を予測することができます。また、FlashAttention を使用してモデル学習を高速化し、MMSeqs2 などの代替 MSA 生成ツールとの互換性を実現しています。
構造を予測する際、OpenFold はまず AlphaFold Evoformer と類似した MSA Transformer Module の複数の層を通過させることにより、タンパク質配列の数値表現を構築します。これにより、MSA 表現 (進化情報を取得)とペア表現 (アミノ酸間の相互作用を取得) 間で交換される情報量が最大化されます。OpenFold は、これらの表現をモジュールを通して複数回再利用することで改善していきます。既定された数のサイクルが終了すると、追加の構造モジュールにおいて個々の残基を中心としたフレームに特殊な形の Attention が適用され、三次元構造を構築および拡張していきます。
タンパク質のフォールディングと設計のための AWS Batch アーキテクチャ
タンパク質のフォールディングと設計のための AWS Batch アーキテクチャは、AWS 上でのタンパク質構造解析のための拡張可能なソリューションです。このソリューションにより、お客様は OpenFold および AlphaFold (訳注参照) で未知のアミノ酸配列の構造を同時に予測することができます。今後、アルゴリズムのサポートを追加していく予定です。お客様は、AWS CloudFormation を使用して、30 分以内にご自身の AWS アカウントにインフラストラクチャをプロビジョニングすることができます。また、UniRef90 や BFD、PDB70 などの依存データを、パブリックリポジトリからセキュアな Amazon FSx for Lustre ファイルシステムに自動的にダウンロードすることが可能です。
[訳注] 2022 年 10 月 時点では、これに加えて OmegaFold もサポートされています。
AWS CloudFormation テンプレートは、必要なネットワークやファイルシステム、コンテナ、およびコンピューティングリソースをプロビジョニングします。また、リクエストに応じて必要なパブリックリファレンスデータをダウンロードします。インストールが完了すると、ユーザーは同梱の BatchFold Python ライブラリを使用して、1つまたは複数の Batch キューにタンパク質の構造解析ジョブを投入することができます。お客様は、提供されたコードを使用して、Amazon SageMaker Studio や AWS Step Functions などの他の AWS サービスからフォールディングジョブを送信することができます。
OpenFold を使用した単量体フォールディング
OpenFold を使った単量体のフォールディングを説明するために、まずは 7FCC のターゲットである IL-1 結合ドメインを作成するところから始めましょう。
import batchfold
target = batchfold.BatchFoldTarget(target_id="7FCC")
target.add_sequence(
seq_id="7FCC_1",
seq="KEYDI...LPLRS",
description="Isoform 4 of IL-1 receptor accessory protein",
)次に、AlphaFold や OpenFold の高精度な解析に必要な MSA ファイルを生成するための JackHMMER ジョブを定義し投入します。今回は、16vCPU・31GiB のシステムメモリで実行されるように指定しました。そして、Graviton-Spot のジョブキューにジョブを投入することで、m6g・r6g・c6gファミリーの中から最適なインスタンスタイプが選択されます。
jackhmmer_job = batchfold.JackhmmerJob(
job_name="My_JackHMMER_Job",
target_id=target.target_id,
fasta_s3_uri=target.get_fasta_s3_uri(),
output_s3_uri=target.get_msas_s3_uri(),
cpu=16, memory=31,
)
jackhmmer_submission = batch_environment.submit_job(
jackhmmer_job, job_queue_name="GravitonSpotJobQueue"
)最後に、ターゲットタンパク質の構造を予測する OpenFold のジョブを定義し投入します。1 GPU を指定し、G4dn のジョブキューにジョブを投入します。depends_on 値を追加することで、OpenFold ジョブは MSA ジョブが正常に終了されるのを待ってから開始されます。
openfold_job = batchfold.OpenFoldJob(
job_name="My_OpenFold_Job",
target_id=target.target_id,
fasta_s3_uri=target.get_fasta_s3_uri(),
msa_s3_uri=target.get_msas_s3_uri(),
output_s3_uri=target.get_predictions_s3_uri() + "/MyJob",
cpu=4, memory=15, gpu=1,
)
openfold_submission = batch_environment.submit_job(
openfold_job, job_queue_name="G4dnJobQueue",
depends_on=[jackhmmer_submission]
)ジョブが完了したら、Amazon Simple Storage Service (Amazon S3) から結果をダウンロードし、py3Dmol を使って構造を可視化します。

図 2 – IL-1 結合ドメインである 7FCC の三次元構造のリボン図。図中の色は、モデルによって割り当てられた、予測局所距離差検定 (pLDDT) によって測定された残基ごとの信頼度スコアを表しています。
構造予測における OpenFold と AlphaFold 2 の比較
AWS Batch 上での OpenFold と AlphaFold の性能を比較するために、2022 年 7 月と 8 月に CAMEO タンパク質ターゲットデータセットに提出された 32 の単量体タンパク質を調査しました。我々は、完全な BFD データベースに対して JackHMMER を使用して各ターゲットの MSA を事前に計算しました。その後、それぞれのアルゴリズムを用いて、デフォルトのパラメータ値のもとで各ターゲットの構造を予測しました。フォールディングジョブは 4 つの vCPU、16GiB のメモリ、および 1 つのT4 GPU を持つ g4dn.xlarge インスタンス上で実行されました。
RCSB Protein Data Bank に登録されている実験的に決定された構造と予測結果を比較し、TMScore を用いて予測精度 (GDT_TS) を計算しました。2つのモデル間の平均 GDT_TS の差は 1% 未満でした。また、19 個のターゲットに対するスコアは 0.9 以上であり、実験レベルの精度を示しました。
AWS Batch は未使用のインスタンスを自動的に終了させるため、各ジョブの総コストは実行時間に大きく依存します。平均して、OpenFold は AlphaFold よりも 90% 速く予測結果を生成しました。これは幾つかの理由によるものです。
- AlphaFold はデフォルトで 5 つのモデルを直列で実行し、最終的な予測を生成します。
- AlphaFold のアルゴリズムでは、各ジョブの開始時にコンパイルステップを含み、事前に学習されたネットワークを最適化します。OpenFold の場合、g4dn のインスタンスタイプではそのような事前コンパイルは必要ありません。
- AWS Batch Architecture for Protein Folding で定義された OpenFold コンテナイメージは、G 系 および P 系の EC2 インスタンスタイプで実行されるように最適化されています。
このデータから、1,300 残基未満の一本鎖タンパク質の構造予測には G4dn のジョブキューで OpenFold を実行することを推奨します。
Amazon EC2 G5 インスタンスによる大規模なタンパク質の解析
1,300 残基以上のタンパク質ターゲットの場合は、以下のステップを推奨します。
- OpenFold のジョブをオプションの G5 ジョブキュー に投入します。これにより、利用可能な VRAM が 16GiB から 24GiB に増加します。G5 インスタンスタイプは、すべての AWS リージョンで利用可能ではないことに注意してください。
- OpenFold ジョブで long_sequence_inference (LSI) フラグを True に設定します。これにより、実行時間が長くなる代わりにメモリ使用量を改善します。この場合、メモリ不足のエラーを防ぐために追加のCPUメモリが必要になる場合があることに注意してください。
LSI を有効化した g4dn.xlarge インスタンスで実行した場合、OpenFold は 1,838 残基のタンパク質の予測を生成しました。この長さは、g5.xlarge インスタンスでは 2,194 残基まで増加しました。
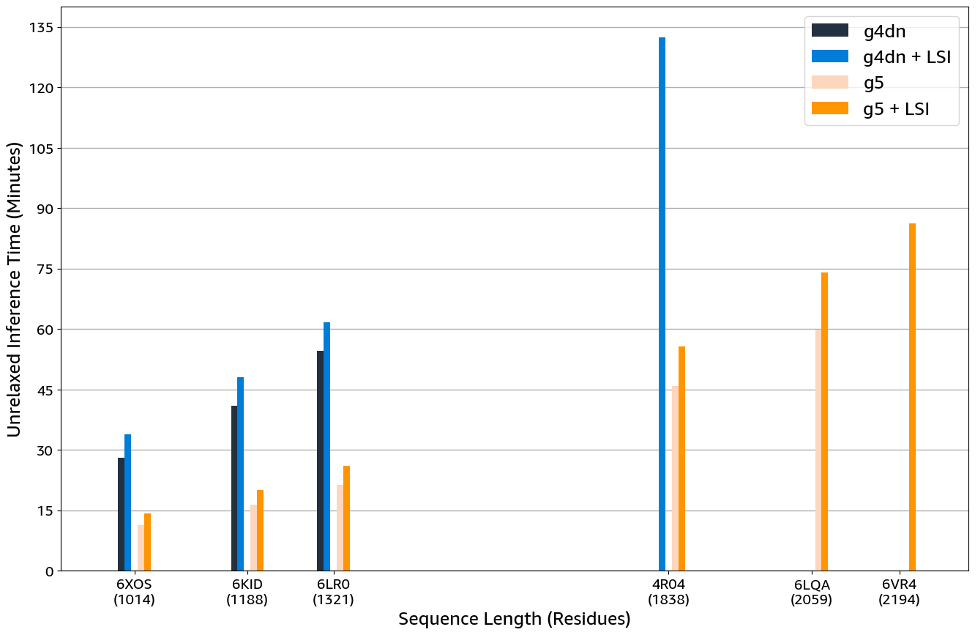
図5 – g4dn および g5 のインスタンスタイプを用いて、OpenFold で巨大タンパク質を予測するために必要な推論時間の比較 (Long Sequence Inference オプションを使用した場合と使用しない場合)。
AWS Graviton2 と Spot インスタンスタイプの使用による MSA 生成のコスト削減
OpenFold と AlphaFold の両方において、エンドツーエンドの予測パイプラインで最も長いステップは MSA 生成です。前回のブログでは、AWS Batch を使用して CPU で MSA を生成することでコスト削減する方法を紹介しました。しかし、Graviton2 ベースのインスタンスやスポットインスタンスを利用することで、さらにコストを削減することが可能です。
AWS Batch Architecture に含まれる MSA 生成コンテナを、デフォルトで Graviton2 プロセッサで実行するように最適化しました。その結果、現行世代の x86 ベースのインスタンスと比較して、最大で 40% の費用対効果の向上が実現しました。さらに、JackHMMER MSA のジョブを Graviton-Spot ジョブキュー に投入することで、最大 90% のコスト削減が可能になります。
7FCC の例では、バッチは JackHMMER ジョブ用に r6g.4xlarge インスタンス (16vCPU、128GiB メモリ )をプロビジョニングし、98 分間実行されました。この記事の執筆時点では、米国東部 (北バージニア) リージョンにおけるこのインスタンスサイズの オンデマンド価格は、1 時間あたり $0.81 です。しかし、スポット価格はわずか $0.30 で、62% の節約となりました。実際のインスタンスタイプとスポット価格は、各リージョンの利用状況によって異なることに注意してください。
最も高価なオプションは、1 つの EC2 インスタンスで両方のジョブを実行することです。しかし、(1) 高速コンピューティングではないインスタンスで別の MSA 生成ジョブを実行、(2) 構造予測に AlphaFold ではなく OpenFold を使用、(3) Graviton2 ベースのインスタンスタイプで MSA 生成ジョブを実行、(4) スポットインスタンスを使用して最大 90% のコスト削減をすることで実行コストを削減することが可能です。
クリーンアップ
進行中の課金をすべて停止するには、まず CloudFormation コンソールに移動し、サイドバーから [スタック] を選択します。次に、[ネスト表示] オプションをオフにして、ネストされたスタックを隠します。最後に、スタックを選択し、[削除] をクリックします。これで、「タンパク質のフォールディングと設計のための AWS Batch アーキテクチャ」に関連するすべてのリソースとデータがアカウントから削除されます。
まとめ
タンパク質構造予測のようなライフサイエンスの問題への機械学習の応用は急速に進んでいます。OpenFold のような新しいアルゴリズムは、研究開発コストを抑制しつつ発見のスピードを向上させることに寄与します。そして AWS Batch のようなフレキシブルな HPC サービスを活用することで、カスタマーは AI 駆動型ツールを必要な時に必要なだけ活用することができるようになります。
著者について
Sachin Kadyan
Sachin Kadyan は、OpenFold のリード開発者です。
Brian Loyal
Brian Loyal は、Amazon Web Services の Global Healthcare and Life Sciences チームのシニア AI/ML ソリューションアーキテクトです。バイオテクノロジーと機械学習の分野で 17 年以上の経験を持ち、お客様がゲノムやプロテオミクスの課題を解決するための支援に情熱を注いでいます。休日は友人や家族と一緒に料理や食事を楽しんでいます。
翻訳は Solutions Architect 森下裕介が担当しました。原文はこちらです。