医療画像のアノテーションとセグメンテーションは、困難な作業ですが深層学習 (DL) 技術によって部分的に自動化することができます。こうした手法は、画素レベルで画像を分類することを目的とする一般的なセグメンテーションタスクにおいて最先端の結果を達成しています。
このブログ記事のパート 1 では、Amazon SageMaker を使用する簡単で合理的な方法で、MRI スキャンから脳組織を自動的にセグメント化するためにニューラルネットワークをトレーニングおよびデプロイする方法を紹介します。Apache MXNet を使って、「Bring Your Own Script」のパラダイムを採用する Amazon SageMaker で畳み込みニューラルネットワーク (CNN) をトレーニングします。U-Net と効率的で低レイテンシーの ENet の 2 つのネットワークをトレーニングします。パート 2 では、AWS Greengrass ML Inference を使用して、低接続または非接続の環境でのオフライン推論用のポータブルエッジデバイスに ENet をデプロイする方法を見ていきます。
この記事ではこのアプローチを脳の MRI に適用しますが、一般的なセグメンテーションの手法として、X 線の分析などの類似のユースケースに適用できます。
このブログ記事では、高レベルの概要を紹介します。完全なチュートリアルのノートブックについては、GitHub にある Amazon SageMaker の脳セグメンテーションを参照してください。
このブログ記事の最後で、ここに示すように MRI からの脳組織のセグメンテーションを予測します。

このユースケースでは、医療画像を保護医療情報 (PHI) ではなく未処理画像として取り扱っていますが、以下の点に注意してください。
AWS Greengrass は、この記事の執筆時点では AWS HIPAA 準拠サービスではありません。AWS Business Associate Addendum (BAA) に従って、AWS Greengrass は、米国の医療保険の携行と責任に関する法律 (HIPAA) における保護医療情報 (PHI) の作成、受信、維持、送信に使用してはなりません。HIPAA の対象であるかどうかを判断する責任はお客様にあります。対象である場合は、HIPAA とその実施規則をどのように遵守するのが最適であるかを判断する必要があります。HIPAA 準拠サービスを使用して PHI を作成、受信、維持、送信するアカウントは、BAA での必要性に応じて PHI を暗号化する必要があります。HIPAA 準拠サービスの最新のリスト、および一般的な詳細については、AWS HIPAA コンプライアンスのページを参照してください。
概要
ユースケース
医療イメージング技術は、医療専門家が人体の内部を見ることを可能にしますが、多くの場合、分析手順および推論のために画像内の組織の正確なセグメンテーションが必要になります。これは特に、患者の心臓血管の健康状態を評価するなどの未処理の画像から洞察を得るために、容積測定および表面分析が重要なユースケースに関係します。通常は、医療専門家がこのセグメンテーションを手作業で行うため、時間がかかります。最近、畳み込みニューラルネットワークが一般的なセグメンテーションタスクにおいて高い性能を示すことが示されています。この記事では、Amazon SageMaker を使用して、MRI 画像から脳組織を自動的にセグメント化する 2 つのネットワークをトレーニングし、デプロイします。Amazon SageMaker は、開発者やデータサイエンティストがあらゆる規模の機械学習 (ML) モデルを迅速かつ簡単に構築、トレーニング、デプロイできるようにする完全マネージド型プラットフォームです。
そうしたモデルでさらに関心を集めるのは、エッジデプロイメントです。エッジでオフラインの推論を実行することは、医療画像のアノテーションに大きな影響を与える可能性があります。インターネットの接続が限られているか、接続がない世界各地では医療専門家が不足しているため、アノテーションをローカルで自動化できるポータブルな低電力ソリューションには多くの利点があります。また、この記事では、AWS Greengrass を使用して Amazon SageMaker でトレーニングしたモデルをエッジにデプロイする方法も示します。このサービスを使用すると、接続されたデバイスのローカルなコンピューティング、メッセージング、データキャッシング、同期、ML 推論の機能を安全に実行できます。
設定
ここで概説した手順を再現するには、このブログ記事に付随するリポジトリにある関連コードとともに Jupyter ノートブックを実行する必要があります。Amazon SageMaker は、ML および DL フレームワークが事前にロードされていて、Jupyter ノートブックを実行する完全マネージド型インスタンスを提供します。リポジトリを起動して、ノートブックインスタンスにリポジトリを複製して、フォローすることを推奨いたします。
注意 – Amazon SageMaker ノートブックインスタンスを使用している場合は、この記事で使用しているデータを別のデバイスに格納する必要があります。可能な選択肢としては、共有メモリの使用、Amazon Elastic File System のマウントがあります。
CNN アーキテクチャ
この記事では、脳組織セグメンテーションのタスクに次の 2 つのモデルを適用します。
データ
この記事では、Open Access Series of Imaging Studies (OASIS) の脳 MRI 断面データの小さなサブセットを使用します。このプロジェクトは、豊富な神経画像データセットを提供します。
探索的データ分析と前処理
ネットワークのトレーニングを開始する前に、データを視覚化してよりよく理解し、セグメンテーションネットワークをトレーニングするために前処理する必要があります。そして、モデルのトレーニング中に Amazon SageMaker が使用できるように、前処理したデータを Amazon S3 に配置します。
視覚化
データは、.img 形式のベクトル画像として提供され、各ファイルは特定の脳の完全な 3 次元 MRI スキャンを表しています。臓器組織のセグメンテーションは本質的に 3 次元のタスクですが、2 次元断面の MRI スライスを分割することで近似します。これにより、体積セグメンテーションよりも複雑さと計算量が少なくなり、適切に機能します。
まず、サンプルを数値配列としてメモリに読み込み、スライスを可視化します。
image = np.fromfile(
open(os.path.join(‘example.img'), 'rb'),
np.dtype('>u2')).reshape((176, 208, 176))
plt.figure(figsize=(12, 12))
plt.title('Input')
plt.imshow(image[:, :, 101], cmap=plt.cm.gray)
メモリに読み込んだ配列は、期待通りに 3 次元です。次の図に示すように、3 番目の軸に沿って特定のスライスを選択しました。

セマンティックセグメンテーションタスクの場合と同様に、各入力のラベルは、それぞれが特定のクラスに一意に対応する整数のマスクです。この場合、これらのクラスは、ここに示すように脳組織と背景のタイプです。
segmentation = np.fromfile(
open(os.path.join(‘example_mask.img'), 'rb'),
np.dtype('>u2')).reshape((176, 208, 88))
plt.figure(figsize=(12, 12))
plt.title('Ground Truth')
plt.imshow(segmentation[:, :, 50], cmap=plt.cm.gray)

前処理
ここで、ネットワークをトレーニングするためにデータを前処理する必要があります。患者によってトレーニングと検証セットにデータを分割 (80/20) します。これは、データの漏れ、つまり隣接する脳 MRI スライスが相関する可能性を排除するために重要であり 、患者による分割によってクリーンな検証を保証します。
train_images, validation_images, train_masks, validation_masks = train_test_split(
images, masks, test_size=0.2, random_state=1984)
MRI 画像を前処理するためには、ネイティブフォーマットから PNG 画像として保存できる配列に変換する必要があります。未処理の MRI 配列は、放射線の強度を表す値を有しています。これらの値はピクセル強度よりもはるかに範囲が広く、uint16 としてメモリに読み込まれます。スライスを PNG として保存すると、データは [0,255] の範囲に収まる uint8 にスケーリングされます。
注意 – このスケーリングは、画像毎の組織のピクセル分布で差異をもたらしますが、これはモデルのトレーニングには影響を与えません。
def process_mris(files, target_dir):
for f in files:
mris = np.fromfile(open(f, 'rb'), dtype='>u2')\
.reshape((176, 208, 176))[:, :, np.arange(1, 176, 2)].transpose((2, 0, 1))
for i, mri in enumerate(mris):
new_fname = "_".join(os.path.basename(f).split('.')[0].split('_')[:8])+"_%i.png" % i
if np.max(mri) <= 255:
imageio.imsave(os.path.join(
target_dir, new_fname), mri.astype(np.uint8))
else:
imageio.imsave(os.path.join(target_dir, new_fname), mri)
return
すべての他のスライスを捉えていることに注意してください。これは、未処理の MRI が対応するマスクの 2 倍の粒度を有するためです。
マスクについては、未処理のマスクを組織クラスごとに正確に 1 ピクセルの画像として保存する必要があります。未処理の画像のピクセル値が正確でないと、それぞれの未処理のマスクのピクセル値は特定の整数にマッピングされます。
def bin_mask(raw_segmentation):
raw_segmentation[raw_segmentation <= 150] = 0
raw_segmentation[np.where((150 < raw_segmentation)
& (raw_segmentation <= 400))] = 1
raw_segmentation[np.where((400 < raw_segmentation)
& (raw_segmentation <= 625))] = 2
raw_segmentation[raw_segmentation > 625] = 3
return raw_segmentation
def process_labels(files, target_dir):
for f in files:
tmp = np.fromfile(open(f, 'rb'), dtype='>u2').reshape(
(176, 208, 88)).transpose((2, 0, 1))
masks = bin_mask(tmp)
for i, mask in enumerate(masks):
new_fname = "_".join(os.path.basename(f).split(
'.')[0].split('_')[:8])+"_%i_mask.png" % i
imageio.imsave(os.path.join(target_dir, new_fname),
mask.astype(np.uint8))
return
ここで、テストサンプルと分散トレーニングのサブセットを作成し、各サブセットを圧縮することができます。
モデルのトレーニング
設定
まず、Amazon SageMaker Python SDK をインポートします。以下を定義します。
- Amazon SageMaker と自分のアカウントのコンテキスト内で便利なメソッドを提供するセッションオブジェクト。
- トレーニングおよびホスティングサービスにアクセス許可を委任するために使用される、Amazon SageMaker のロール ARN。これらのサービスが、データとモデルが保存されている Amazon S3 バケットにアクセスできるために、これが必要です。
import sagemaker
from sagemaker import get_execution_role
from sagemaker.mxnet import MXNet
sagemaker_session = sagemaker.Session()
role = get_execution_role()
また、Amazon SageMaker のエスティメーターオブジェクト MXNet もインポートしました。このオブジェクトが AWS が提供するデフォルトのコンテナを参照するので、ユーザーはエントリポイントスクリプトとサポートコードだけを提供します。このチュートリアルでは MXNet フレームワークを使用していますが、Tensorflow、Chainer、さらに現在は PyTorch を使用して独自のスクリプトを簡単に導入することもできます。
Amazon S3 へのデータのアップロード
次に、upload_data メソッドを使用して前処理されたデータを Amazon S3 にアップロードし、デフォルトの Amazon SageMaker バケットにオブジェクトを配置します。圧縮ファイルを含んでいる親ディレクトリをメソッドに渡すと、このメソッドが子ツリーをバケットに同期します。
prefix = 'brain-segmentation-tar-gz'
data_bucket = sagemaker_session.upload_data(path=tar_gz_dir, key_prefix=prefix)
エントリポイント
前述のように、MXNet エスティメーターがデフォルトの MXNet コンテナを使用し、ユーザーは単にトレーニングを定義するコードを提供します。(たとえば、GitHub にある Apache MXNet モジュール API を使用した SageMaker モデルのトレーニングとホスティングを参照してください。)
エントリポイントとしてエスティメーターに渡すスクリプトは、brain_segmentation.py です。
import os
import tarfile
import mxnet as mx
import numpy as np
from iterator import DataLoaderIter
from losses_and_metrics import avg_dice_coef_metric
from models import build_unet, build_enet
import logging
logging.getLogger().setLevel(logging.DEBUG)
###############################
### トレーニングループ ###
###############################
def train(current_host, channel_input_dirs, hyperparameters, hosts, num_cpus, num_gpus):
logging.info(mx.__version__)
# インスタンス環境に基づいてコンピューティングのコンテキストを設定する
if num_gpus > 0:
ctx = [mx.gpu(i) for i in range(num_gpus)]
else:
ctx = mx.cpu()
# Set location of key-value store based on training config.
if len(hosts) == 1:
kvstore = 'device' if num_gpus > 0 else 'local'
else:
kvstore = 'dist_device_sync' if num_gpus > 0 else 'dist_sync'
# ハイパーパラメータを取得する
batch_size = hyperparameters.get('batch_size', 16)
learning_rate = hyperparameters.get('lr', 1E-3)
beta1 = hyperparameters.get('beta1', 0.9)
beta2 = hyperparameters.get('beta2', 0.99)
epochs = hyperparameters.get('epochs', 100)
num_workers = hyperparameters.get('num_workers', 6)
num_classes = hyperparameters.get('num_classes', 4)
class_weights = hyperparameters.get(
'class_weights', [[1.35, 17.18, 8.29, 12.42]])
class_weights = np.array(class_weights)
network = hyperparameters.get('network', 'unet')
assert network == 'unet' or network == 'enet', '"network" hyperparameter must be one of ["unet", "enet"]'
# 圧縮されたトレーニング/検証データを見つける
train_dir = channel_input_dirs['train']
validation_dir = channel_input_dirs['test']
train_tars = os.listdir(train_dir)
validation_tars = os.listdir(validation_dir)
# 圧縮されたイメージ/マスクのペアをローカルに抽出する
for train_tar in train_tars:
with tarfile.open(os.path.join(train_dir, train_tar), 'r:gz') as f:
f.extractall(train_dir)
for validation_tar in validation_tars:
with tarfile.open(os.path.join(validation_dir, validation_tar), 'r:gz') as f:
f.extractall(validation_dir)
# 抽出したデータの場所にカスタムイテレータを定義する
train_iter = DataLoaderIter(
train_dir,
num_classes,
batch_size,
True,
num_workers)
validation_iter = DataLoaderIter(
validation_dir,
num_classes,
batch_size,
False,
num_workers)
# ネットワークのシンボルグラフを構築する
if network == 'unet':
sym = build_unet(num_classes, class_weights=class_weights)
else:
sym = build_enet(inp_dims=train_iter.provide_data[0][1][1:], num_classes=num_classes, class_weights=class_weights)
logging.info("Sym loaded")
# グラフをモジュールに読み込む
net = mx.mod.Module(sym, context=ctx, data_names=('data',), label_names=('label',))
# カスタムメトリクスを初期化する
dice_metric = mx.metric.CustomMetric(feval=avg_dice_coef_metric, allow_extra_outputs=True)
logging.info("Starting model fit")
# モデルのトレーニングを開始する
net.fit(
train_data=train_iter,
eval_data=validation_iter,
eval_metric=dice_metric,
initializer=mx.initializer.Xavier(magnitude=6),
optimizer='adam',
optimizer_params={
'learning_rate': learning_rate,
'beta1': beta1,
'beta2': beta2},
num_epoch=epochs)
# パラメータを保存する
net.save_params('params')
# 推論のみのグラフを構築し、トレーニングモデルからパラメータを設定する
if network == 'unet':
sym = build_unet(num_classes, inference=True)
else:
sym = build_enet(
inp_dims=train_iter.provide_data[0][1][1:], num_classes=num_classes, inference=True)
net = mx.mod.Module(
sym, context=ctx, data_names=(
'data',), label_names=None)
# バッチサイズ 1 のモデルを再バインドする
net.bind(data_shapes=[('data', (1,) + train_iter.provide_data[0][1][1:])])
net.load_params('params')
return net
このスクリプトは、トレーニングジョブのライフサイクルを構成するトレーニングループを定義します。注意するべきポイント:
- ハイパーパラメータ – このハイパーパラメータのマッピングをトレーニング設定の一部としてエスティメーターに渡します。
- ネットワーク – このハイパーパラメータは、unet または enet のどちらがトレーニングするネットワークであるかを定義します。
- ローカルインポート – モデル、イテレータ、およびロス/メトリクスの定義はローカルモジュールにあります。このスクリプトと付随するモジュールをエスティメーターに渡します。
チュートリアルリポジトリのモジュールを参照して、これらのモデルやイテレータの詳細な情報を得ることをお勧めします。
注意 – ML ハイパーパラメータのチューニングは複雑で時間がかかり、専門知識が必要です。ニューラルネットワークの場合、これはさらに真実です。Amazon SageMaker の自動モデルチューニングにより、開発者やデータサイエンティストは、ベイジアン最適化を使用して特定のメトリクスで最適化するハイパーパラメータチューニングジョブを起動できます。このモデルを含む Amazon SageMaker のトレーニングジョブで、自動モデルチューニングを使用できます。方法を確認するには、GitHub にある Amazon SageMaker および MXNet によるハイパーパラメータチューニングから始めることをお勧めします。
ローカルテストにローカルモードを使用する
自分のスクリプトやモデルを Amazon SageMaker に導入する場合は、コードをテストしてバグがないことを確認する必要があります。ベストプラクティスは、カスタムアルゴリズムのコンテナをローカルでテストすることです。 そうしない場合は、Amazon SageMaker がトレーニングインスタンスを起動して、エラーを受け取るまで待つ必要があります。これは、ローカルモードでサポートされています。
コンテナをローカルで実行するには、Amazon SageMaker でローカルで実行する Docker をダウンロード、インストール、設定します。これは、このヘルパースクリプトで実行できます。
次に、Amazon S3 の入力タイプを定義します (サンプルセットでテストします)。
sample_train_s3 = sagemaker.s3_input(s3_data=os.path.join(
data_bucket, "sample-train"), distribution='FullyReplicated')
sample_validation_s3 = sagemaker.s3_input(s3_data=os.path.join(
data_bucket, "sample-validation"), distribution='FullyReplicated')
これで、エントリポイントのスクリプトや付随するコードのディレクトリなどを含めて、トレーニング設定を使ってエスティメーターを定義することができます。train_instance_type を local に設定して、ジョブをローカルで起動することに注意してください。
local_unet_job = 'DEMO-local-unet-job-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
local_estimator = MXNet(entry_point='brain_segmentation.py',
base_job_name=local_unet_job,
source_dir='source_dir',
role=role,
train_instance_count=1,
train_instance_type='local',
hyperparameters={
'learning_rate': 1E-3 * 16,
'class_weights': [[1.35, 17.18, 8.29, 12.42]],
'network': 'unet',
'batch_size': 8,
'epochs': 1
})
ここで、Amazon S3 入力で fit を呼び出し、 train および test のラベルを互いにマッピングします。ローカルモードなので、コンテナがダウンロードされ、イメージはローカルで実行されます。
local_estimator.fit({'train': sample_train_s3, 'test': sample_validation_s3})
次に、deploy を呼び出して、サービスイメージをローカルでもテストします。
local_unet_endpoint = 'DEMO-local-unet-endpoint-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
local_predictor = local_estimator.deploy(initial_instance_count=1, instance_type='local',
endpoint_name=local_unet_endpoint)
deploy メソッドは、エンドポイントにリクエストを送信するために使用できる predictor を返します。
response = local_predictor.predict(test_brain.tolist())
output = np.argmax(np.array(response), axis=(1))[0].astype(np.uint8)
plt.figure(figsize=(14, 14))
plt.subplot(131)
plt.title('Input')
plt.imshow(test_brain[0][0], cmap=plt.cm.gray)
plt.subplot(132)
plt.title('Ground Truth')
plt.imshow(test_mask[0][0], cmap=plt.cm.gray)
plt.subplot(133)
plt.title('Prediction')
plt.imshow(output, cmap=plt.cm.gray)
サンプルデータセットの 1 つのエポックでは、次の図に示すように、結果は酷いものになると予測されます。より多くのエポックに対してトレーニングすると、パフォーマンスが向上します。
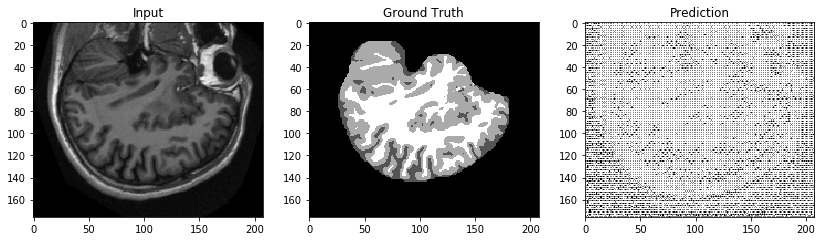
スクリプトをテストした結果、自信を持って実際のトレーニングジョブとエンドポイントを起動することができます。
U-Net
まず、より高速で軽量な ENet と比較するためのベースラインとして U-Net をトレーニングします。デフォルトでは、トレーニングジョブは 100 エポックに対して実行されます。
unet_single_machine_job = 'DEMO-unet-single-machine-job-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
unet_single_machine_estimator = MXNet(entry_point='brain_segmentation.py',
base_job_name=unet_single_machine_job,
source_dir='source_dir',
role=role,
train_instance_count=1,
train_instance_type='ml.p3.2xlarge',
hyperparameters={
'learning_rate': 1E-3,
'class_weights': [[1.35, 17.18, 8.29, 12.42]],
'network': 'unet',
'batch_size': 32,
})
今回は、トレーニングセット全体をトレーニングし、検証セット全体を検証します。
train_s3 = sagemaker.s3_input(s3_data=os.path.join(
data_bucket, "train"), distribution='FullyReplicated')
validation_s3 = sagemaker.s3_input(s3_data=os.path.join(
data_bucket, "validation"), distribution='FullyReplicated')
unet_single_machine_estimator.fit({'train': train_s3, 'test': validation_s3})
ENet
ENet をトレーニングするには、network ハイパーパラメータを enet に変更するだけです。
enet_single_machine_job = 'DEMO-enet-single-machine-job-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
enet_single_machine_estimator = MXNet(entry_point='brain_segmentation.py',
base_job_name=enet_single_machine_job,
source_dir='source_dir',
role=role,
train_instance_count=1,
train_instance_type='ml.p3.2xlarge',
hyperparameters={
'learning_rate': 1E-3,
'class_weights': [[1.35, 17.18, 8.29, 12.42]],
'network': 'enet',
'batch_size': 32,
})
enet_single_machine_estimator.fit({'train': train_s3, 'test': validation_s3})
分散トレーニング
私たちがトレーニングを行ったデータは、OASISで利用できるデータのごく一部です。大規模なトレーニングでは、時間とコストの両方で、分散トレーニングがネットワークをトレーニングするためのコスト効率の高いアプローチであることがよくあります。ここでの規模では、このアプローチの速さは単一のマシンとあまり変わらないかもしれませんが、デモとして提供しています。
Amazon SageMaker で独自のスクリプトを使用して分散トレーニングのジョブを開始するには、次のようにわずかのステップだけで十分です。
- 提供するスクリプトが、分散トレーニングをサポートしている必要があります。MXNet では、キーバリューストアを定義してこれを行います。エントリポイントのスクリプトのこの部分は、ジョブに複数のデバイスがあるかどうかを確認し、複数のデバイスがある場合は、同期分散トレーニングのキーバリューストアを定義します。
if len(hosts) == 1:
kvstore = 'device' if num_gpus > 0 else 'local'
else :
kvstore = 'dist_device_sync' if num_gpus > 0 else 'dist_sync'
train_instance_count を 1 から複数に設定します。- ノード間でトレーニングデータをシャードします。
最後のステップでは、distribution を ShardedByS3Key に設定して、シャードした Amazon S3 入力を定義します。このオプションは、指定されたプレフィックスの下にあるオブジェクトの数をノード間で均等に分割しようとします (これは暗黙のうちに、プレフィックスの下にノードと同様に多くのオブジェクトが必要であることを意味します)。この引数を FullyReplicated として保存していた場合、各ノードにはデータセット全体のコピーがあります。
distributed_train_s3 = sagemaker.s3_input(s3_data=os.path.join(
data_bucket, "dist"), distribution='ShardedByS3Key')
これで、分散トレーニングジョブを起動できます。
unet_distributed_job = 'DEMO-unet-distributed-job-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
unet_distributed_estimator = MXNet(entry_point='brain_segmentation.py',
base_job_name=unet_distributed_job,
source_dir='source_dir',
role=role,
train_instance_count=2,
train_instance_type='ml.p3.2xlarge',
hyperparameters={
'epochs': 50,
'learning_rate': 1E-3,
'class_weights': [[1.35, 17.18, 8.29, 12.42]],
'network': 'unet',
'batch_size': 32,
})
unet_distributed_estimator.fit({'train': distributed_train_s3,
'test': validation_s3})
推論エンドポイントのデプロイ
両方のモデルをトレーニングしたので、Amazon SageMaker を使用してウェブでホストされる HTTP エンドポイントにデプロイし、低レイテンシーで推論と予測に役立てることができます。デフォルトでは、これらのエンドポイントはメモリ内のイメージ配列の POST リクエストに応答します。
Deploying the U-Net エンドポイントのデプロイ
まず、エンドポイントに U-Net をデプロイします。
unet_endpoint = 'DEMO-unet-endpoint-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
unet_predictor = unet_single_machine_estimator.deploy(instance_type='ml.c5.xlarge',
initial_instance_count=1, endpoint_name=unet_endpoint)
テストレスポンスを取得して、推論を入力およびグラウンドトゥルースと比較してみましょう。
response = unet_predictor.predict(test_brain.tolist())
output = np.argmax(np.array(response), axis=(1))[0].astype(np.uint8)
plt.figure(figsize=(14, 14))
plt.subplot(131)
plt.title('Input')
plt.imshow(test_brain[0][0], cmap=plt.cm.gray)
plt.subplot(132)
plt.title('Ground Truth')
plt.imshow(test_mask[0][0], cmap=plt.cm.gray)
plt.subplot(133)
plt.title('Prediction')
plt.imshow(output, cmap=plt.cm.gray)
定性的に言えば、これらの結果からは以下の画像が有望に見えます。
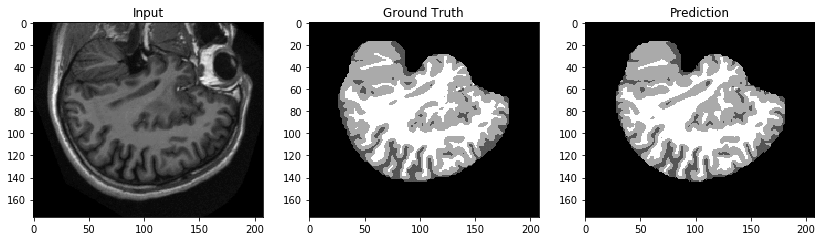
ENet エンドポイントのデプロイ
次に、ENet モデルをデプロイします。エスティメーターで deploy メソッドを使用する代わりに、演習として、モデルオブジェクト MXNetModel を作成してみます。トレーニングジョブから、ENet モデルの成果物の場所を渡します。これは、Amazon SageMaker に独自のモデルを導入してエンドポイントにデプロイする方法です。
enet_endpoint = 'DEMO-enet-endpoint-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
enet_model = sagemaker.mxnet.MXNetModel(enet_single_machine_estimator.model_data, role,
'brain_segmentation.py', source_dir='source_dir', framework_version="1.0")
enet_predictor = enet_model.deploy(
instance_type='ml.c5.xlarge', initial_instance_count=1)
テストレスポンスを取得して、推論を入力およびグラウンドトゥルースと比較してみましょう。
response = enet_predictor.predict(test_brain.tolist())
output = np.argmax(np.array(response), axis=(1))[0].astype(np.uint8)
plt.figure(figsize=(14, 14))
plt.subplot(131)
plt.title('Input')
plt.imshow(test_brain[0][0], cmap=plt.cm.gray)
plt.subplot(132)
plt.title('Ground Truth')
plt.imshow(test_mask[0][0], cmap=plt.cm.gray)
plt.subplot(133)
plt.title('Prediction')
plt.imshow(output, cmap=plt.cm.gray)
再び、これらの結果からは以下の画像が有望に見えます。

Amazon S3 URI を使用してエンドポイントを呼び出す
Amazon SageMaker の MXNet エンドポイントは、デフォルトでは POST リクエスト経由で送信されたデータの推論を実行するように設定されています。送信されるデータは、モデルがトレーニングされたデータの形式であると仮定します。コンピュータビジョンのモデルの場合、これが Amazon SageMaker が要求する帯域幅制限のために問題になる可能性があります。
1 つの回避策は、代わりに Amazon S3 Uniform Resource Identifier (URI) を送信し、エンドポイントに画像をローカルでダウンロードさせて変換し、結果のマスクを Amazon S3 に戻すことです。このアプローチでは、変換されるコンテンツのサイズに制限はありません。
それでは、このカスタム配信画像のエントリポイントがどのように見えるかを調べましょう。
from __future__ import absolute_import
import boto3
import base64
import json
import io
import os
import mxnet as mx
from mxnet import nd
import numpy as np
mx.test_utils.download("https://s3.amazonaws.com/sagemaker-png/png.py", "png.py")
import png
###############################
### ホスティングコード ###
###############################
def push_to_s3(img, bucket, prefix):
"""
画像の配列を png 形式でエンコードし、Amazon S3 にプッシュするメソッド
パラメータ:
----------
img : np.array
アップロードする画像を表す整数の配列。
bucket : str
アップロードする先の S3 バケット。
prefix : str
エンコードされた画像をアップロードするプレフィックス (.png にする必要があります)。
"""
s3 = boto3.client('s3')
png.from_array(img.astype(np.uint8), 'L').save('img.png')
response = s3.put_object(
Body=open('img.png', 'rb'),
Bucket=bucket,
Key=prefix
)
return
def download_from_s3(bucket, prefix):
"""
Amazon S3 からオブジェクトをダウンロードするメソッド
パラメータ:
----------
bucket : str
アップロードする元の Amazon S3 バケット。
prefix : str
ダウンロードする元のプレフィックス 。
"""
s3 = boto3.client('s3')
response = s3.get_object(Bucket=bucket, Key=prefix)
return response
def decode_response(response):
"""
Amazon S3 呼び出しからの未処理画像のバイトを mx.ndarray へデコードするメソッド。
パラメータ:
----------
response : dict
Amazon S3 get_object 応答のディクショナリ。
"""
data = response['Body'].read()
b64_bytes = base64.b64encode(data)
b64_string = b64_bytes.decode()
return mx.image.imdecode(base64.b64decode(b64_string)).astype(np.float32)
def transform_fn(net, data, input_content_type, output_content_type):
try:
inp = json.loads(json.loads(data)[0])
bucket = inp['bucket']
prefix = inp['prefix']
s3_response = download_from_s3(bucket, prefix)
img = decode_response(s3_response)
img = nd.expand_dims(nd.transpose(img, (2, 0, 1)), 0)
img = nd.sum_axis(nd.array([[[[0.3]], [[0.59]], [[0.11]]]]) * img, 1, keepdims=True)
batch = mx.io.DataBatch([img])
net.forward(batch)
raw_output = net.get_outputs()[0].asnumpy()
mask = np.argmax(raw_output, axis=(1))[0].astype(np.uint8)
output_prefix = os.path.join(
'output', '/'.join(prefix.split('/')[1:]).split('.')[0] + '_MASK_PREDICTION.png')
push_to_s3(mask, bucket, output_prefix)
response = {'bucket': bucket, 'prefix': output_prefix}
except Exception as e:
response = {'Error': str(e)}
return json.dumps(response), output_content_type
すでにトレーニングしたモデルがあるので、以前のトレーニングジョブからのモデル成果物の Amazon S3 ロケーションで MXNetModel を使用して、直接モデルオブジェクトを定義します。
unet_s3_endpoint = 'DEMO-unet-s3-endpoint-' + \
time.strftime("%Y-%m-%d-%H-%M-%S", time.gmtime())
unet_s3_model = sagemaker.mxnet.MXNetModel(unet_single_machine_estimator.model_data, role,
entry_point="brain_segmentation_s3_transform.py", source_dir='source_dir')
unet_s3_predictor = unet_s3_model.deploy(instance_type='ml.c5.xlarge', initial_instance_count=1,
endpoint_name=unet_s3_endpoint)
ここで、Amazon S3 にテスト画像をアップロードします。
s3 = boto3.client('s3')
test_bucket = "<YOUR-BUCKET-HERE>"
prefix = "test_img.png"
s3.upload_file(Filename=os.path.join(‘test.png'),
Bucket=test_bucket, Key=prefix)
リクエストの本文は JSON 形式であるため、関連するフィールドを含むディクショナリを定義し、エンドポイントを呼び出します。
request_body = json.dumps({"bucket": test_bucket, "prefix": prefix})
response = unet_s3_predictor.predict([request_body])
ここで結果をダウンロードし、視覚化します。
s3.download_file(Bucket=response['bucket'],
Key=response['prefix'], Filename="result.png")
plt.figure(figsize=(14, 14))
plt.subplot(131)
plt.title('Input')
plt.imshow(test_brain[0][0], cmap=plt.cm.gray)
plt.subplot(132)
plt.title('Ground Truth')
plt.imshow(test_mask[0][0], cmap=plt.cm.gray)
plt.subplot(133)
plt.title('Prediction')
plt.imshow(np.array(Image.open("result.png")), cmap=plt.cm.gray)

モデルの評価と比較
どちらのモデルも質的に優れていますが、これを数値化して 2 つのモデル間でレイテンシーを比較する必要があります。ENet は、効率と低レイテンシーのために精度をトレードオフするように設計されていることを思い出してください。
混同行列の使用
マルチクラス分類のパフォーマンスを視覚化するための効果的な方法は、グラウンドトゥルースラベルと予測ラベルを比較する混同行列です。セマンティックセグメンテーションは、表向きはピクセルごとのマルチクラス分類であるため、ここでも混同行列が便利です。
混同行列をプロットするために、scikit-learn ドキュメントの以下のコードを使用します。
from sklearn.metrics import confusion_matrix
import itertools
def plot_confusion_matrix(cm, classes,
normalize=False,
title='Confusion matrix',
cmap=plt.cm.Blues):
"""
この関数は、混同行列を出力し、プロットします。
「normalize = True」と設定することで、正規化を適用できます。
"""
if normalize:
cm = cm.astype('float') / cm.sum(axis=1)[:, np.newaxis]
print("Normalized confusion matrix")
else:
print('Confusion matrix, without normalization')
print(cm)
plt.imshow(cm, interpolation='nearest', cmap=cmap)
plt.title(title)
plt.colorbar()
tick_marks = np.arange(len(classes))
plt.xticks(tick_marks, classes, rotation=45)
plt.yticks(tick_marks, classes)
fmt = '.2f' if normalize else 'd'
thresh = cm.max() / 2.
for i, j in itertools.product(range(cm.shape[0]), range(cm.shape[1])):
plt.text(j, i, format(cm[i, j], fmt),
horizontalalignment="center",
color="white" if cm[i, j] > thresh else "black")
plt.tight_layout()
plt.ylabel('True label')
plt.xlabel('Predicted label')
#Citations
#@article{scikit-learn,
# title={Scikit-learn: Machine Learning in {P}ython},
# author={Pedregosa, F. and Varoquaux, G. and Gramfort, A. and Michel, V.
# and Thirion, B. and Grisel, O. and Blondel, M. and Prettenhofer, P.
# and Weiss, R. and Dubourg, V. and Vanderplas, J. and Passos, A. and
# Cournapeau, D. and Brucher, M. and Perrot, M. and Duchesnay, E.},
# journal={Journal of Machine Learning Research},
# volume={12},
# pages={2825--2830},
# year={2011}
#}
U-Net エンドポイントを使用して、検証セットからのグラウンドトゥルースと比較する予測を得ることができます。
ground_truths = []
outputs = []
for validation_pair in validation_pairs:
img = np.array(Image.open(os.path.join(validation_dir, validation_pair[0])))[
np.newaxis, np.newaxis, :]
response = unet_predictor.predict(img.tolist())
outputs.append(np.argmax(np.array(response), axis=(1))[0].astype(np.uint8))
ground_truths.append(
np.array(Image.open(os.path.join(validation_dir, validation_pair[1]))))
ground_truths = np.concatenate(ground_truths)
outputs = np.concatenate(outputs)
ground_truths = ground_truths.flatten()
outputs = outputs.flatten()
これで、予測をグラウンドトゥルースラベルと比較することができます。
# 混同行列を計算する
cnf_matrix = confusion_matrix(ground_truths, outputs)
np.set_printoptions(precision=2)
class_names = [0, 1, 2, 3]
# 非正規化混同行列をプロットする
plt.figure()
plot_confusion_matrix(cnf_matrix, classes=class_names,
title='Confusion matrix, without normalization')
# 正規化混同行列をプロットする
plt.figure()
plot_confusion_matrix(cnf_matrix, classes=class_names, normalize=True,
title='Normalized confusion matrix')
plt.show()
これにより、次の図に示すように、正規化された混同行列が得られます。
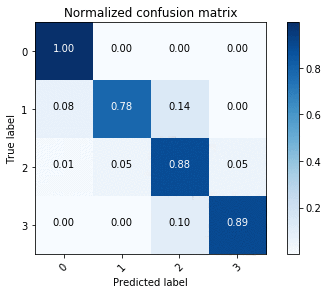
ENet エンドポイントで同じことを行うと、次の画像の混同行列が得られます。
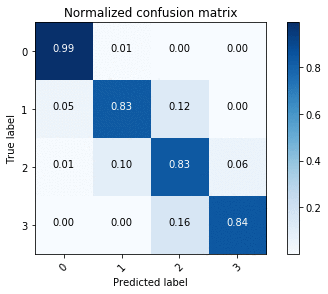
混同行列を使用することで、全体的な精度に関して U-Net が ENet より優れていることがわかりますが、これは精度 – レイテンシーのトレードオフを前提としています。
推論のレイテンシーを定量化する
2 つのモデルの推論時間を定量化するために、次の環境で U-Net と ENet の順方向パスを計算しました。
バッチサイズ 1、8、16、32、64 について試行しました。
CPU 環境での結果
次の図で示すように、すべてのバッチサイズで、ENet は U-Net よりも高速で実行され、リアルタイムです。ENet は、U-Net のようなモデルに比べて FLOPS が 75 倍少なくなるように設計されており、レイテンシーへの影響は明らかです。

GPU 環境での結果
興味深いことに、次の図に示すように、バッチサイズが小さい場合、U-Net が ENet より高速です。
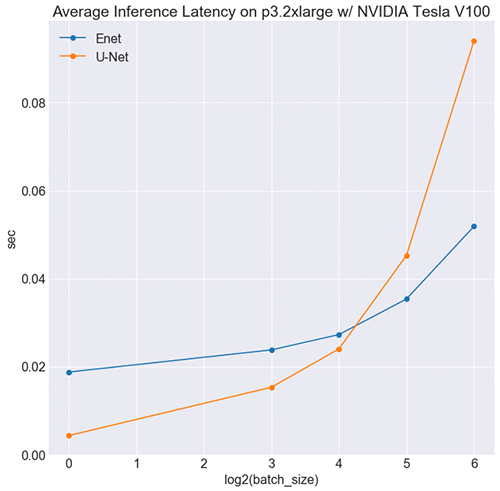
これは直観に反するかもしれませんが、各ネットワークの記号グラフのそれぞれのサイズを考慮すると意味があります。記号的には ENet は U-Net よりもかなり大きく (ここを参照)、推論の際により多くのカーネルを起動します。より小さいバッチサイズでは、カーネルを起動してメモリを転送するコストが ENet の計算に関するメリットを上回り、速度が遅くなります。一定の閾値では、計算効率がこのコストを上回り、U-Net よりも高速です。
とりわけカーネルフュージョンを提供する NVIDIA TensorRT を使って ENet の計算グラフを最適化することで、これを改善することができます。この技術は、レイヤーとテンソルを融合して、各カーネルの起動が確実に計算を最大化するようにします。MXNet にもすぐに導入されます。
RPi (CPU) 環境での結果
CPU でのベンチマーク結果に基づいて、次の図に示すように、ENet は RPi 上で U-Net より優れたパフォーマンスを発揮すると予測しており、それはまさに事実です。
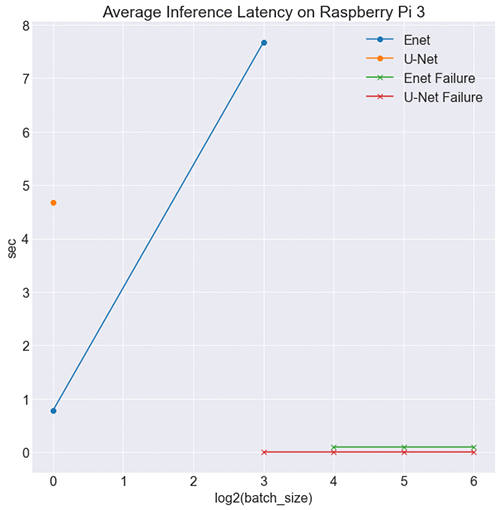
もっと重要なことは、ENet が低リソース環境でどのように効果的であるかということです。バッチサイズが 16、32、64 の場合、RPi はテストデータをメモリに保持できませんでした。ただし、バッチサイズ 8 では、U-Net は失敗しましたが、ENet は失敗しませんでした。これは、RPi ボードが同じバッチサイズの U-Net 推論を処理できなかったため、ENet の影響が小さいことの証です。また、Enet は、U-Net の 30Mb と比較して 1.5Mb のパラメータでモデルサイズの点でも影響が少なく、エッジ推論に理想的です。
まとめ
この記事のパート 1 では、独自の MXNet スクリプトを Amazon SageMaker に導入することで、畳み込みニューラルネットワークをトレーニングして、脳組織のセグメンテーションを自動化する方法を示しました。ローカルモードを使用してスクリプトをローカルでテストする方法と、分散トレーニングジョブ用にデータをシャードする方法を示しました。デフォルトのエンドポイントのデプロイメントとは別に、画像データの代わりに Amazon S3 URI に応答するカスタムサービングコードを記述する方法を示しました。U-Net と ENet の間での精度と効率のトレードオフについて検討し、低リソース環境では ENet が理想的であると判断しました。
パート 2 では、AWS Greengrass を使用して Amazon SageMaker でトレーニングした ENet モデルをエッジデバイスにデプロイする方法を説明します。脳のセグメンテーションのためのポータブル ML ソリューションとしての推論を行います。
謝辞
この作業は、CC BY 4.0 で使用されている「Open Access Series of Imaging Studies (OASIS), OASIS-1, by Marcus et al., 2007」によって提供されたデータで可能になりました。
以下のデータが、OASIS によって提供されました。
- OASIS-3: 主任研究者: T. Benzinger, D. Marcus, J. Morris; NIH P50AG00561, P30NS09857781, P01AG026276, P01AG003991, R01AG043434, UL1TR000448, R01EB009352.AV-45 薬は、Eli Lilly の 100% 子会社である Avid Radiopharmaceuticals によって提供されました。
- OASIS: 断面: 主任研究者: D. Marcus, R, Buckner, J, Csernansky J. Morris; P50 AG05681, P01 AG03991, P01 AG026276, R01 AG021910, P20 MH071616, U24 RR021382.
- OASIS: 縦方向: 主任研究者: D. Marcus, R, Buckner, J. Csernansky, J. Morris; P50 AG05681, P01 AG03991, P01 AG026276, R01 AG021910, P20 MH071616, U24 RR021382.
出版物:
- Open Access Series of Imaging Studies (OASIS): 「Cross-Sectional MRI Data in Young, Middle Aged, Nondemented, and Demented Older Adults」
Marcus, DS、Wang, TH、Parker, J、Csernansky, JG、Morris, JC、Buckner, RL。Journal of Cognitive Neuroscience (認知神経科学ジャーナル)、19, 1498-1507. doi: 10.1162/jocn.2007.19.9.1498
今回のブログ投稿者について
 Brad Kenstler は、Amazon Machine Learning ソリューションラボチームのデータサイエンティストです。ML ソリューションラボの一員として、彼は AWS の顧客が自社ビジネスのユースケースやプロセスのために組織内で機械学習や AI を活用するのを手助けしています。彼の主な関心分野は、コンピュータビジョンと深層学習が交わる領域です。仕事以外では、ヘビーメタルを聴くこと、新しいバーボンを飲むこと、サンフランシスコ 49ers の試合観戦などを楽しんでいます。
Brad Kenstler は、Amazon Machine Learning ソリューションラボチームのデータサイエンティストです。ML ソリューションラボの一員として、彼は AWS の顧客が自社ビジネスのユースケースやプロセスのために組織内で機械学習や AI を活用するのを手助けしています。彼の主な関心分野は、コンピュータビジョンと深層学習が交わる領域です。仕事以外では、ヘビーメタルを聴くこと、新しいバーボンを飲むこと、サンフランシスコ 49ers の試合観戦などを楽しんでいます。
.










 Brad Kenstler は、Amazon Machine Learning ソリューションラボチームのデータサイエンティストです。ML ソリューションラボの一員として、彼は AWS の顧客が自社ビジネスのユースケースやプロセスのために組織内で機械学習や AI を活用するのを手助けしています。彼の主な関心分野は、コンピュータビジョンと深層学習が交わる領域です。仕事以外では、ヘビーメタルを聴くこと、新しいバーボンを飲むこと、サンフランシスコ 49ers の試合観戦などを楽しんでいます。
Brad Kenstler は、Amazon Machine Learning ソリューションラボチームのデータサイエンティストです。ML ソリューションラボの一員として、彼は AWS の顧客が自社ビジネスのユースケースやプロセスのために組織内で機械学習や AI を活用するのを手助けしています。彼の主な関心分野は、コンピュータビジョンと深層学習が交わる領域です。仕事以外では、ヘビーメタルを聴くこと、新しいバーボンを飲むこと、サンフランシスコ 49ers の試合観戦などを楽しんでいます。
